全体を通してやりたいこと
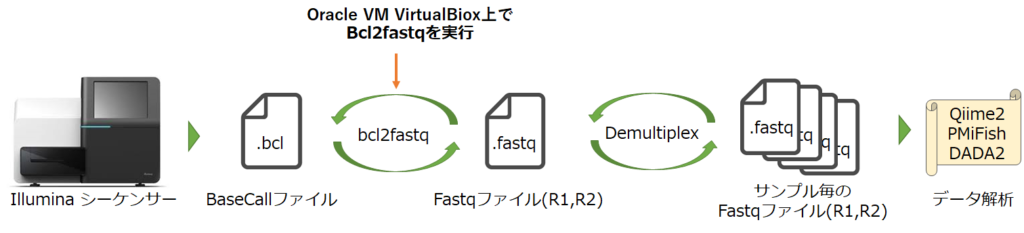
Part1ではBaseSpace Sequence Hub CLIを利用して、ネット経由でRunデータをダウンロードする方法について解説しました。
Part2ではダウンロードしてきたRunファイルを使って、bclファイルからfastqファイルを生成するためのOracle VM VirtualBoxを使った環境構築から、bcl2fastqによるファイル変換までを解説していきたいと思います。
1. LocalでbclファイルからFastqファイルを取得したい理由
Miseqの配列決定は1サイクルごとにATGCそれぞれの塩基で励起させた蛍光を写真撮影して、その情報からBinary Base Call(bcl)ファイルを生成する流れになってます。
サンプルシートにGenerateFASTQをしていると、Illuminaシーケンサーに搭載されているCASAVAによってbcl2fastqが実行されて、.bclファイルから.fastqファイルが生成されます。
そして同時に、解読されたタグ配列部分の情報とサンプルシートに記載されている各サンプルのタグ配列情報を照合することで、どのサンプル由来のアンプリコンなのかを判別して配列を振り分けるdemultiplexが実行されます。
ただ、CASAVAによるdemultiplexはタグ配列部分に付与されているQscore(読み取り信頼スコア)を利用していないらしく、品質の良し悪しに関わらず、決定された塩基配列情報だけを使ってFastqファイルが分割されます。また、塩基配列の不一致も一部許容しているようです。
どのくらいの影響があるのかはわかりませんが、せっかく読み取り信頼度が付与されているのであれば、データのクオリティをあげるためにも使用したほうがいいと思います。
なのであえてIlluminaシーケンサー内で動くbcl2fastqは使わず、local環境でbcl2fastqを実行してdemultiplexされていないファイルを生成後、下流のデータ解析を進めたいと思います。
2. なぜ”windows PCで”なのか
解析用PCを用意されている方は、多分UbuntuなどのLinuxディストリビューショやMacOSが搭載されていることが多いと思います。
一方専用PCがない場合、会社で用意されているのは大抵windows PCでしょう。
2017年のWindows10のアップデート以降、Linux コマンドラインツールを使いたい時は、WSLを使用することで、仮想マシンやデュアルブーストといったwindowsとは全く異なる環境を作成する必要がなくなりました。
環境DNAで扱うようなベーシックな解析では、多分WSLやPowershellといったwindowsの標準機能を利用すれば大抵問題はありません。
しかし、bclファイルをfastqファイルへ変換するbcl2fasqはLinux環境?でしか動作しません。
1度、WSL+Ubuntuでbcl2fastqの実行を試みた時も確かに動作しませんでした。何か逃げ道はあるかもしれませんが、それより独立したLinuxの環境をPC内に構築した方が早いと思ったので、今回はその方針で進めます。
Windows PC内にLinux環境を構築する
WSLがwindows PCの標準機能となる前は、windowsでLinuxを使いたい場合、独立した仮想的なLinux環境を作り上げることでその問題をクリアしていました。
仮想環境で代表的なのは、Oracle VM VirtualBoxが挙げられます。構築や運用が簡単で日本語の解説記事も多いと思います。
簡単な比較については、こちらが分かりやすかったです。
一応、当ブログのデータ解析環境の構築というページにも仮想マシンの設定という見出しでVirtual BoxへXubuntuをインストールする流れを解説していますが、ちょうどPCが変わったので次はVirtual BoxへUbuntuをインストールしていきたいと思います。
Oracle VM VirtualBiox
Oracle VM VirtualBioxをここ(https://www.virtualbox.org/wiki/Downloads)からダウンロードします。
Windows hostsを選択するとインストーラーのダウンロードが始まります(103MB)。ダウンロードが終わったら、インストーラーの指示に従って進めます。
Nextをクリック
Nextをクリック
スタートメニュー、デスクトップ、ツールバーにショートカットが必要であれば、1,2,3にチェックを入れたままにしておきます。最下段は.vboxに関連付けをするかについてです。これはそのままにしておいたほうがいいと思います。
インストール中に一時的にネットワークが切れるかもしれないよという警告です。赤文字はドキッとしますね。
Yesをクリックして進めます。
ここまで来たらあとはInstallをクリックしてインストールを開始します。
Install中…

終わりました。このままFinishをクリックして終了するとOracle VM VirtualBoxマネージャーが自動で開きます。
新規をクリックして、ダイヤログボックスが出てきたらエキスパートモードをクリック。
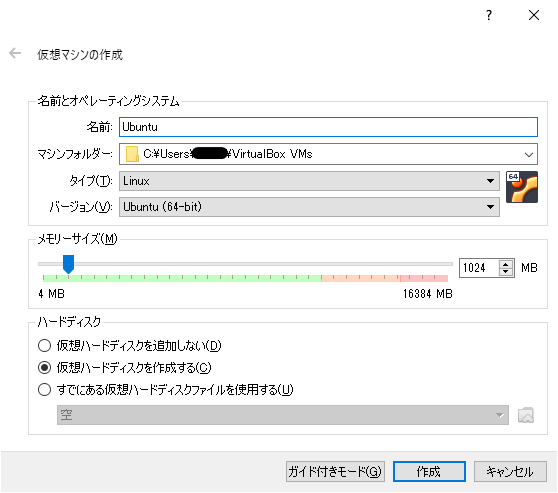
必要事項を記入していきます。
名前はなんでもいいですが、Ubuntuとしておきます。タイプはLinuxでバージョンはUbuntu (64-bit)です。
仮想マシンに割り当てるメモリですが、私のPCは16GBメモリなので最大値が16*1024=16,384MBとなっています。ここは後で変更可能なので、1024MBのまま作成をクリックして進みます。
そうすると先ほど指定したc:\Users\(User名)フォルダ内にUirtualBox VMs というフォルダが作成されています。その中にはUbuntuフォルダがあるはずです。その中にダウンロードしたisoイメージファイルを移動させておきましょう。
次に仮想マシンのストレージタイプやファイルサイズを選択します。

まず可変サイズと固定サイズがありますが、以下のような特徴があります。
- 可変サイズ
設定したサイズの仮想ディスクがホスト側のOSのハードディスクに適用されるのではなく、使用した分だけホストOSのハードディスクを使用する。 - 固定サイズ
設定したサイズの仮想ディスクがホストOSのハードディスクに適用される。
10GBがデフォルトサイズになっていますが、後々変更するのが面倒なので少し多めにとって40GBとしました。
ハードディスクのファイルタイプはVDI(VirtualBox Dis Image)を選択します。
ここまで出来たら作成をクリック。
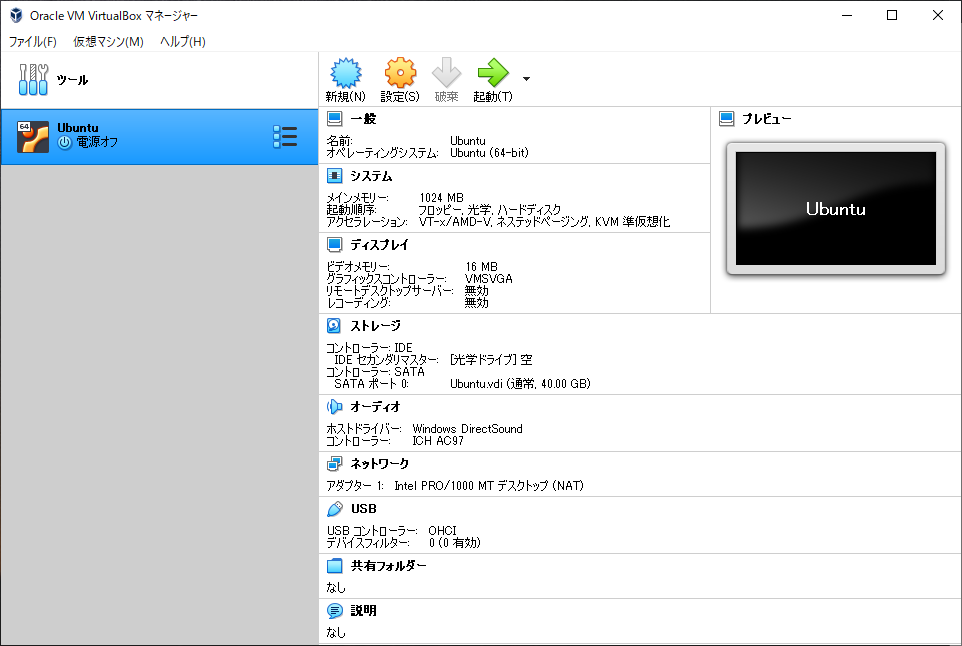
VirtualBoxの起動画面に、作成した仮想マシンが表示されていれば一段落です。
Ubuntu 20.04 LTSのデスクトップイメージファイルのダウンロード

Ubuntu https://ubuntu.com/
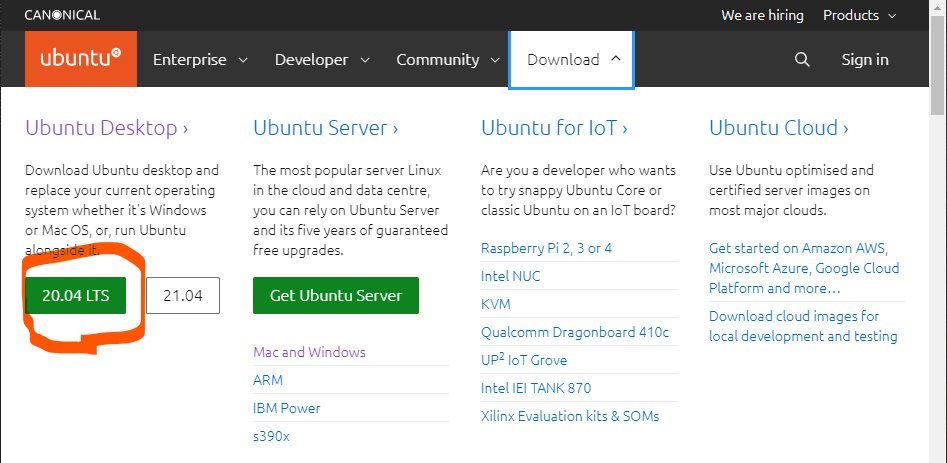
ここから20.04 LTS(Long Term Support:長期サポート)をクリックすると、イメージファイルのダウンロードが開始されます。2.9GBあるので注意してください。
Ubuntu20.04 LTSのインストール
仮想マシンにUbuntu 20.04 LTSをインストールします。物理的なCDはありませんが、CDからドライブをインストールするような感じです。

ス ストレージをクリックして、コントローラーIDEの “空” をクリック。IDEセカンダリマスター横のCDマークをクリックします。
仮想光学ディスクの選択/作成をクリック、追加をクリックして、 ダウンロードフォルダにあるubuntu-20.04.3-desktop-amd64.isoを選択して、選択をクリックします。
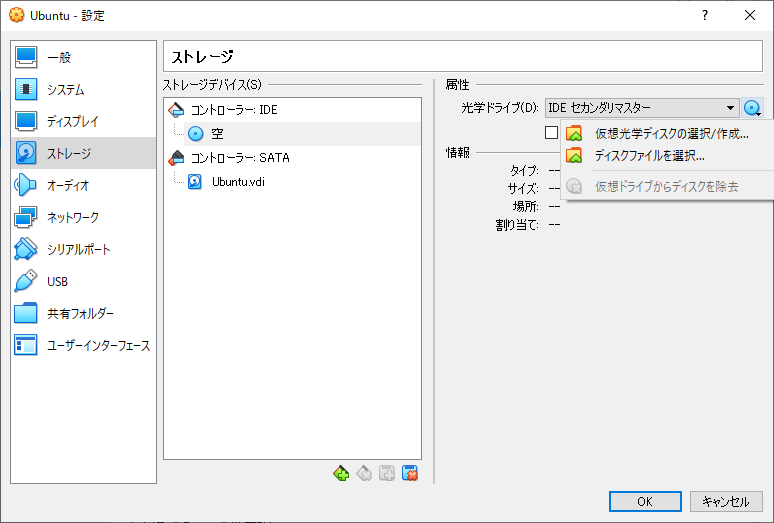
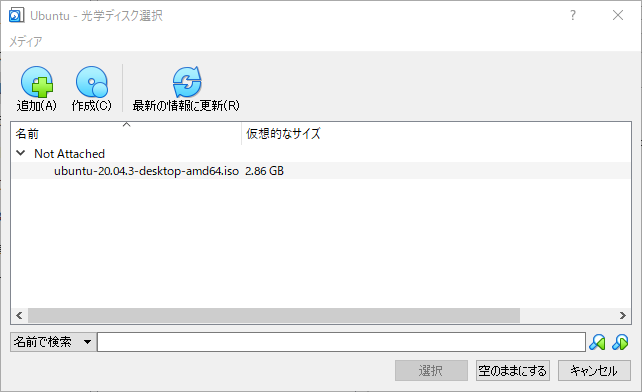
最後にOKを押して準備は完了です。
VirtualBoxによるUbuntuの起動と設定
左の仮想マシンをダブルクリックして起動させます。
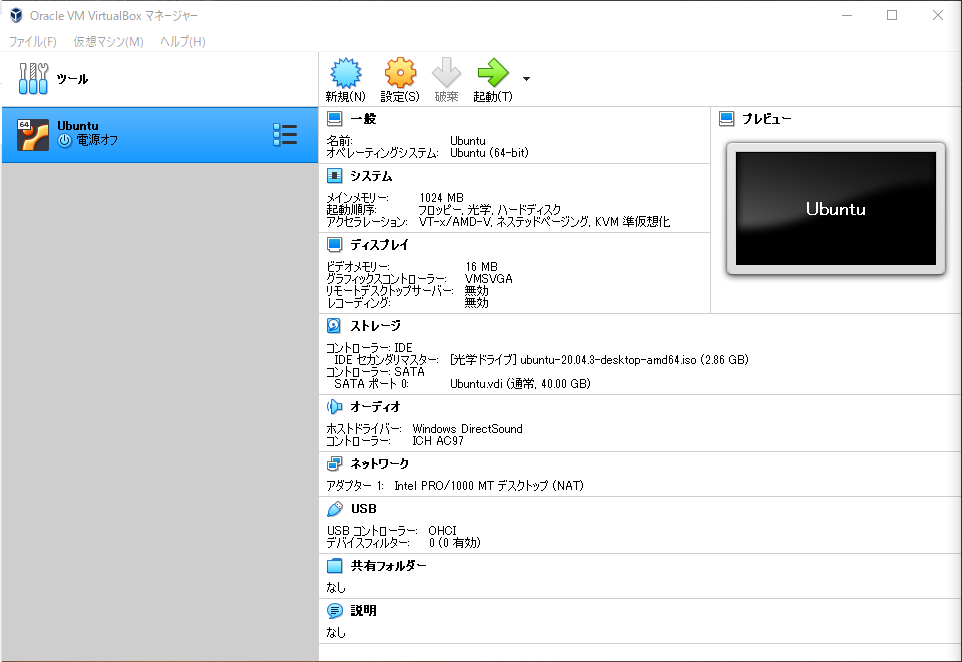
少し待機するとディスクイメージのチェックが終わり、インストーラーが起動します。
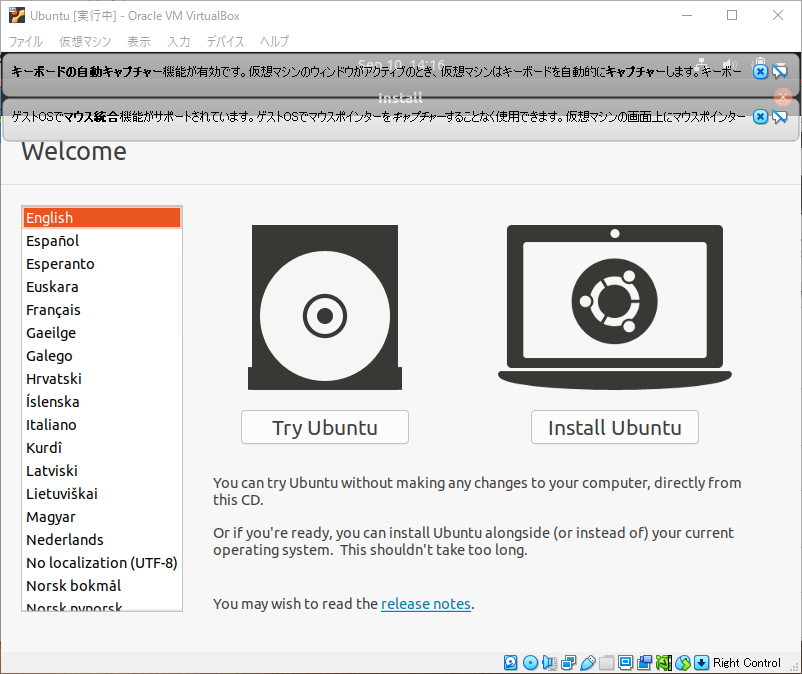
右のInstall Ubuntuをクリックして、Ubuntuをインストールします。

英字キーボードの場合は”English(US)”、一般的なキーボードなら”Japanese”になるので、合うものを選択して”Continue”をクリックします。
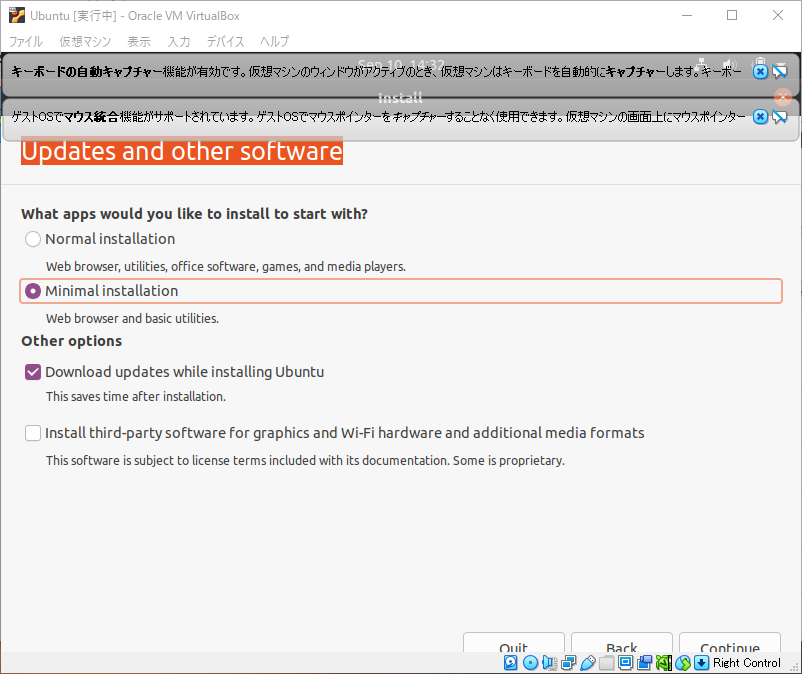
“Updates and other software”は”What apps would you like to install to start with?”はFireFoxなどのwebブラウザが初めから必要なければ”Minimal installation”を選択します。
“Other options”は”Download updates while installing Ubuntu”を選択し、Continueをクリックするとインストールが開始されます。
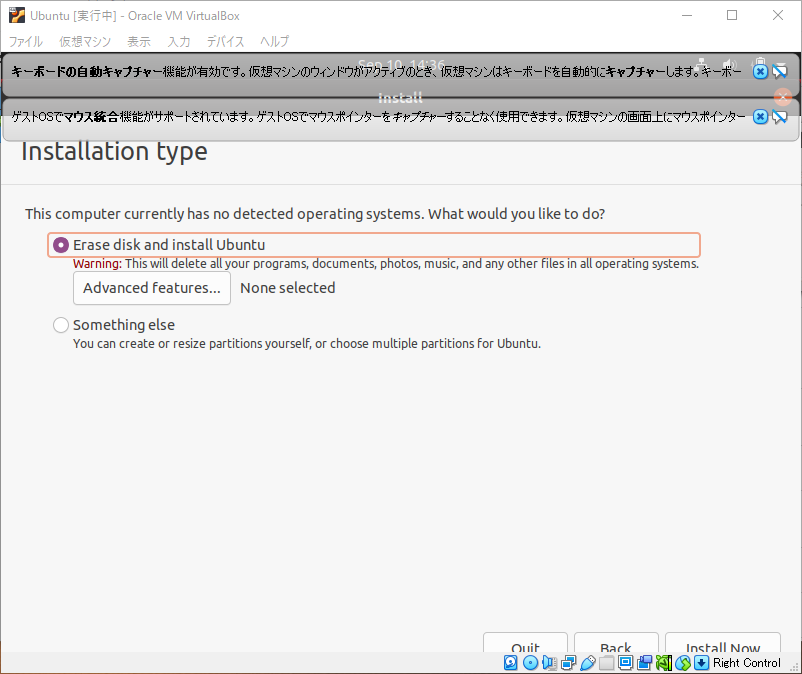
もともとUbuntuが入っていないので、OSが見つかりませんでした。ディスクを削除してUbuntuをインストールしますか?と確認されるので、そのままInstall Nowをクリック。
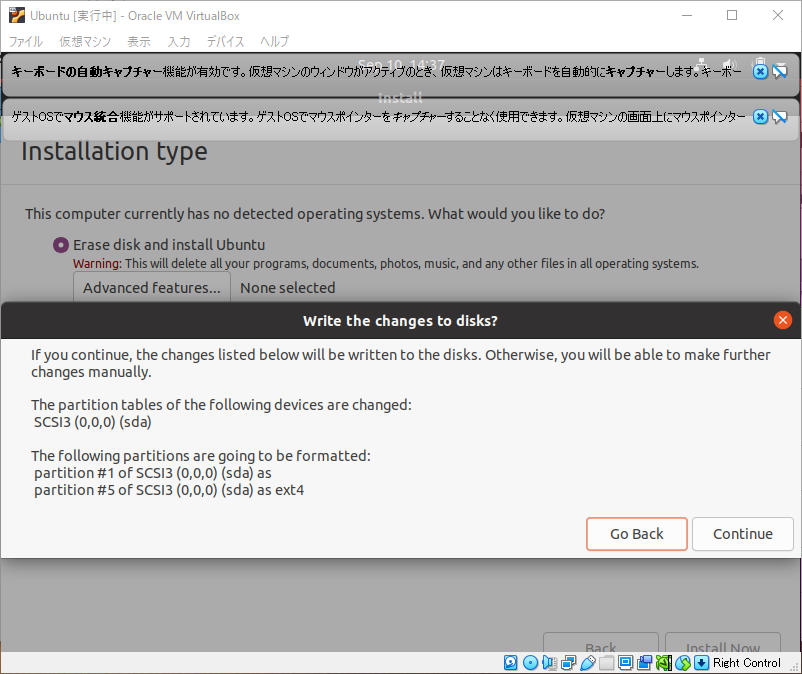
気にせず続けます。Continueをクリック。
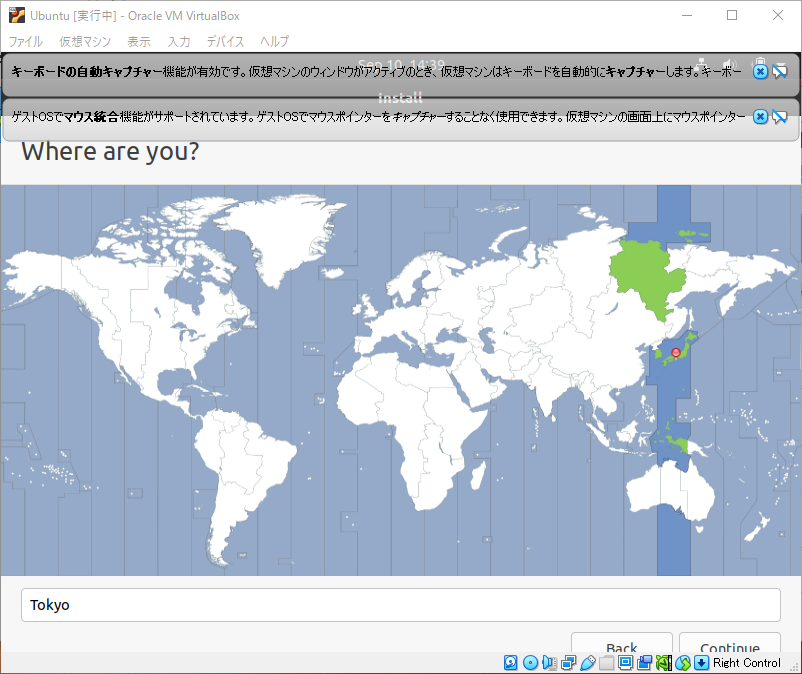
続いて、地域を選択します。日本であれば”Tokyo”を選択して、”続ける”をクリックします。
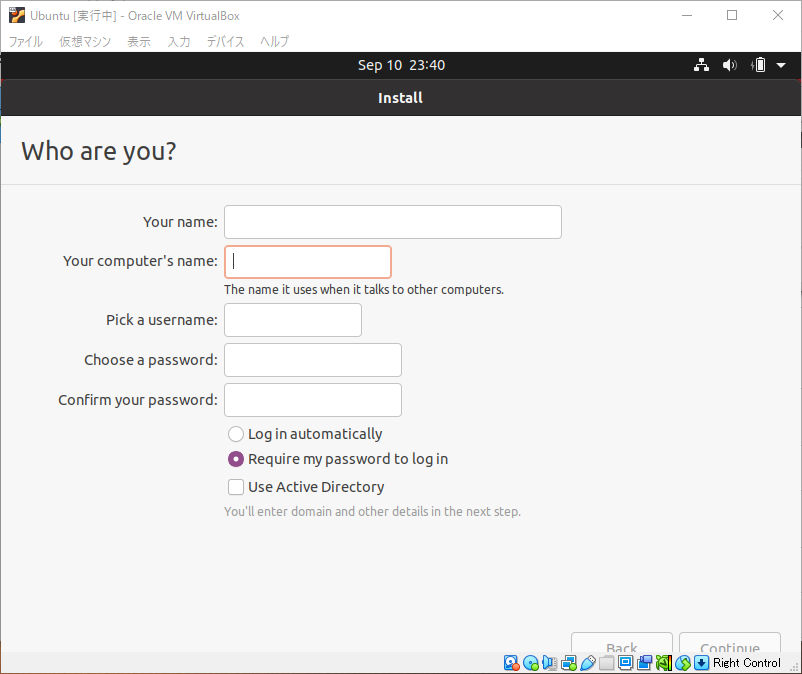
アカウント情報とコンピューターの情報を入力します。すべて半角英数字で記入することをお勧めします。記入し終わったら”続ける”をクリックします。
インストールは少し時間がかかります。 ※ストレージの容量を大きく設定しすぎた場合、この段階でエラーが出たりします。
インストールが終了したら、再起動を求められるので”今すぐ再起動する”をクリックして、仮想環境システムを再起動します。
再起動するとアカウントの選択画面が表示されるので、先ほど設定したパスワードを入力します。 これで、VirtualBoxへのUbuntuのインストールは完了です。
初期設定1 : 解像度を変更する
VirtualBoxにインストールしたUbuntuの画面サイズは640×480で固定されていますが、Guest Additionsのインストールを実行することで、解像度の変更ができるようです。
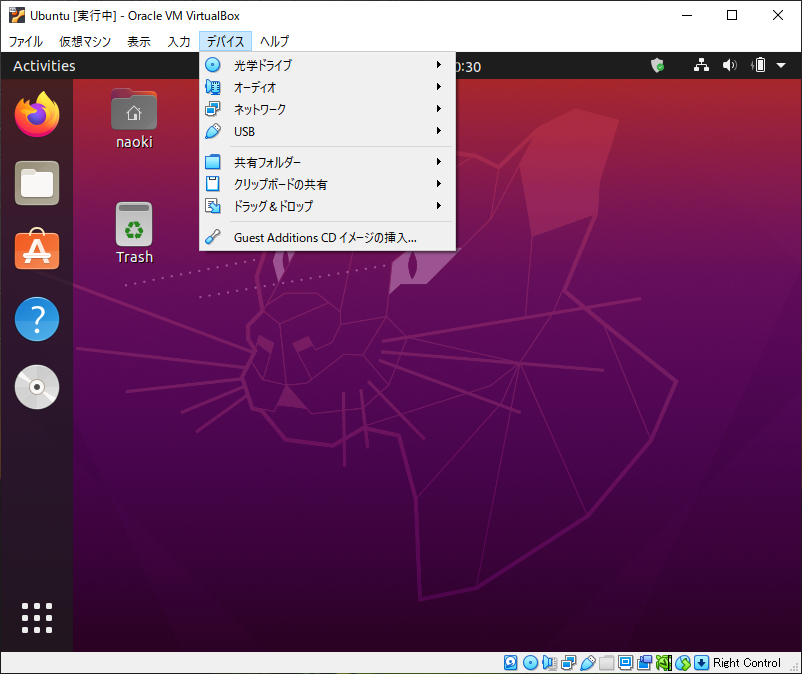
デバイス → Guest Additionsのインストール
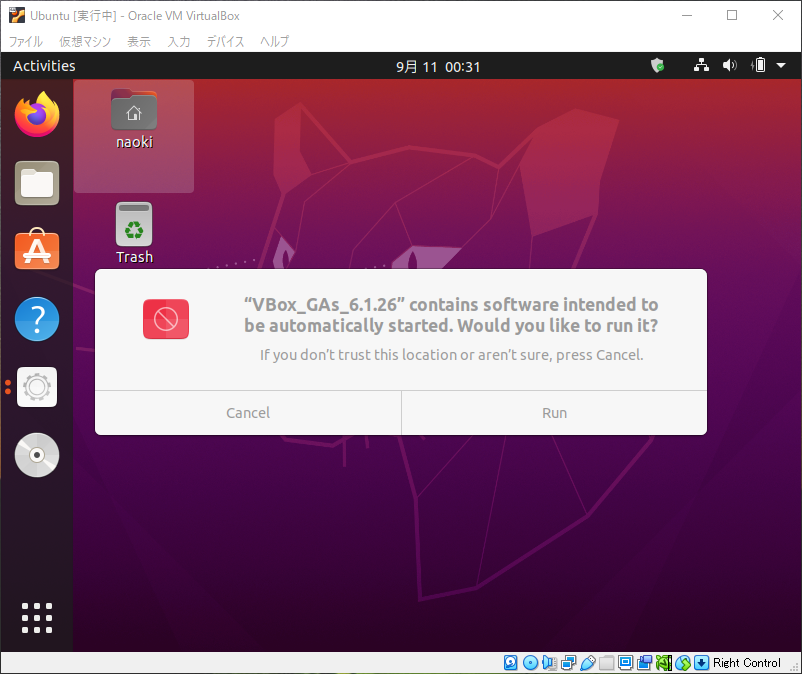
Runを選択して、表示されるダイヤログボックスにパスワードを記入
Press Return to close this window…とコマンドラインに表示されればOK。再起動をかけてると画面サイズに応じてUbuntuの画面サイズが変更されるようになります。
初期設定2 : 仮想マシンとホストOSとの共有フォルダを設定する
仮想マシンとホストOS間でファイル共有ができる共有フォルダの設定をします。この設定をしておくと一部解析を任せている場合に重宝します。
初めにホストOSのデスクトップにUbuntu_hsfというフォルダを作成します。次に仮想マシンのデスクトップにUbuntu_gsfというフォルダを作成します。これらを共有フォルダにすることにします。
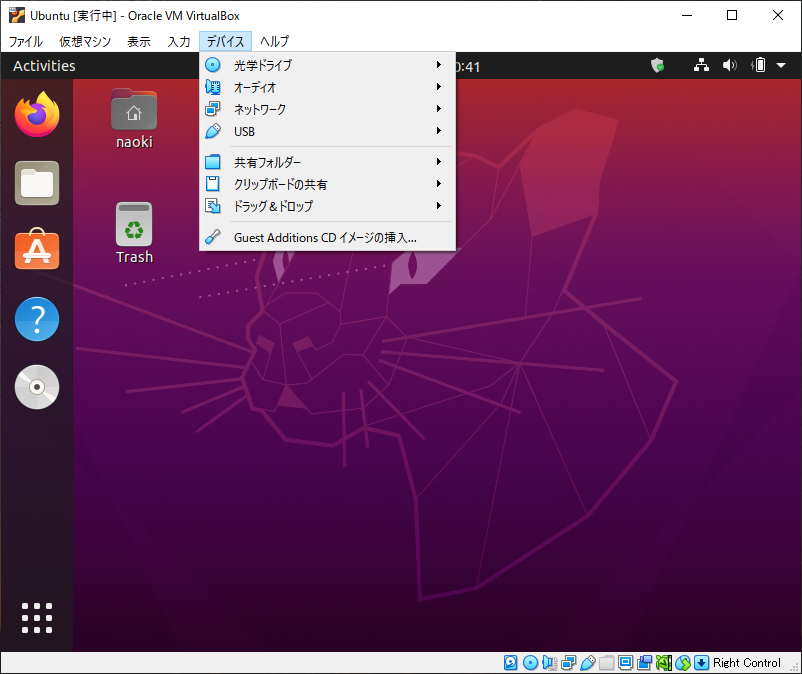
デバイス → 共有フォルダー → 共有フォルダー設定
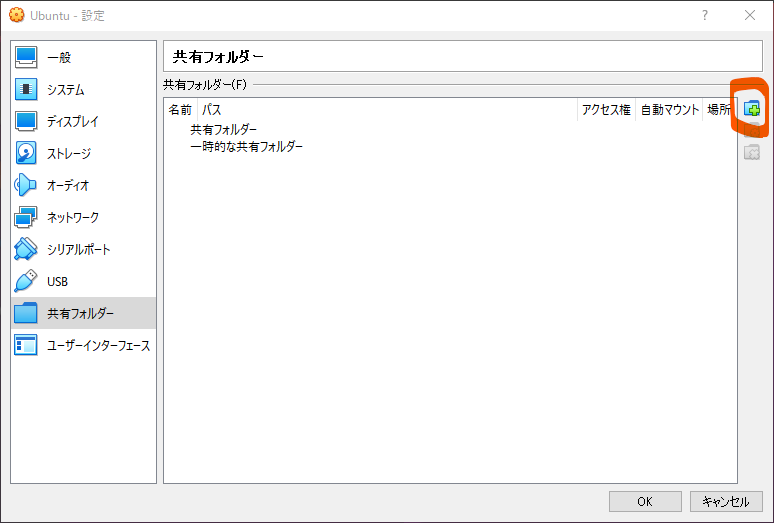
新規共有フォルダを追加をクリック
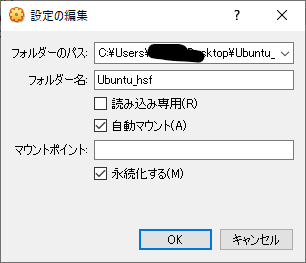
フォルダーのパスを設定して、自動マウントにチェックを入れます。マウスポイントは空白にしておいてOKです。
次にアクセス権限の設定をします。
Oracle VM VirtualBox内でTerminalを開いて下記コードを入力します。
|
1 |
$ sudo gpasswd -a (ユーザー名) vboxsf |
“Adding user (ユーザー名) group vboxsf”と出力されればOKです。次に、フォルダのマウントをします。
Terminal上で、
|
1 |
$ sudo mount -t vboxsf Ubuntu_hsf /home/(ユーザー名)/Desktop/Ubuntu_gsf |
これでホストOSであるWindows上のUbuntu_hsf内に作成したフォルダを仮想マシン上に作成したUbuntu_gsf内に表示・編集することができるようになりました。
初期設定3 : Ubuntu 20.04で日本語入力できるようにする
現状, 仮想マシン上のUbuntu 20.04では日本語入力ができません。よって、Japanese(Mozc)を有効化して、日本語入力ができるようにします。
setting applicationを開き、”Region & Language”を選択します。
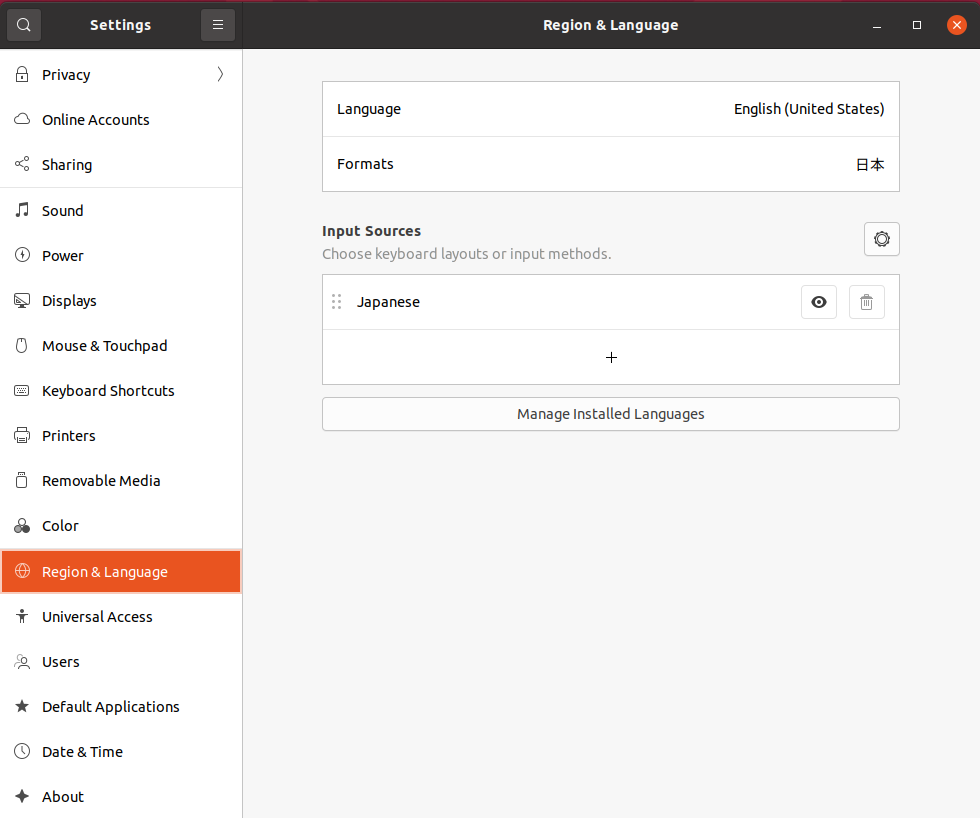
Input Sourcesの”+”をクリックして、”Add an Input Source” ダイヤログボックス内のJapaneseをクリック。
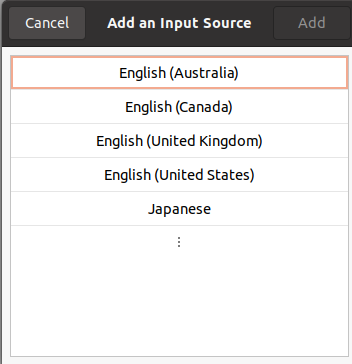
Japanese(Mozc)を選択し、緑色に光る”Add”をクリックします。
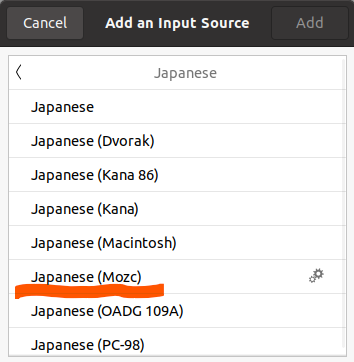
もし、Japanese (Mozc)が候補にない場合は、仮想マシン上のterminalで下記コードを実行してください。
|
1 2 3 |
$ sudo apt install ibus-mozc $ ibus restart $ gsettings set org.gnome.desktop.input-sources sources "[('xkb', 'jp'), ('ibus', 'mozc-jp')]" |
仮想マシン内にbcl2fastqをインストールする
Linux環境が整備できたので、次はbclファイルからFastqファイルを生成するbcl2fastqをインストールします。ここでインストールするbcl2fastqはv2のものです。bcl2fastqはIlluminシーケンサーのReal Time Analyzer(RTA)のバージョンによって使用するバージョンが異なります。
またv1はインストール方法も実行方法も異なりますので注意してください。1度別の用事でインストールしましたがかなり面倒でした。
このパートでは下記3記事を参考にしました。
- DADA2 と Claident を用いた short-read amplicon sequence のデータ解析
(https://ushio-ecology-blog.blogspot.com/2019/11/20191129blogger0002_30.html) - Metabarcoding and DNA barcoding for Ecologists: Sequence analysis
(https://github.com/astanabe/MetabarcodingTextbook/blob/master/metabarcodingtextbook2.ja.pdf) - bcl2fastq2 Conversion Software v2.20
(https://jp.support.illumina.com/content/dam/illumina-support/documents/documentation/software_documentation/bcl2fastq/bcl2fastq2-v2-20-software-guide-15051736-03.pdf)
移行の作業は構築した仮想マシン内で行います。
まず、Terminalを起動します。
ホームディレクトリに移動して下記コマンドを実行していきます。
|
1 2 3 4 5 |
#ホームディレクトリに移動 $ cd # rpm2cのインストール $ sudo apt install rpm2cpio cpio |
|
1 2 |
# bcl2fastq2のダウンロード $ wget -O bcl2fastq2-v2-18-0-12-linux-x86-64.zip http://bit.ly/2drPH3W |
|
1 2 |
# bcl2fastqの解凍 unzip -qq bcl2fastq2-v2-18-0-12-linux-x86-64.zip |
|
1 2 |
# rpmファイルからcpioファイルへの変換 $ rpm2cpio bcl2fastq2-v2.18.0.12-Linux-x86_64.rpm | cpio -id |
|
1 2 3 4 |
# 移動と削除 $ sudo mv usr/local/bin/bcl2fastq /usr/local/bin/ $ sudo cp -R usr/local/share/css /usr/local/share/ $ sudo cp -R usr/local/share/xsl /usr/local/share/ |
これで仮想マシン内にbcl2fastqがインストールされました。
helpコマンドで確認してみます。
|
1 |
$ bcl2fastq -h |
エラーなしでコマンドオプションが表示されればOK
いくつか主要なオプションを見ておきましょう。bcl2fastqの後に続けるオプションコマンドです。
bcl2fastqのオプション
| オプションコマンド | 入力例 | 意味 |
| –processing-threads | 8 | 使用するCPUコア数 |
| –use-bases-mask | Y150n, I8, I8, Y150n | 両側から150 + 1cycleと8bpのインデックス配列を両側から配列解読をした解析を対象とする. nは1サイクルを表す |
| –runfolder-dir | test | “test”という名前のRunフォルダを対象とする |
| –sample-sheet | sample_sheet_edit.csv | “sample_sheet_edit.csv”という名前のsample_sheetファイルを参照する |
| –output-dir | output | “output”という名前のフォルダに解析結果を出力する |
| –create-fastq-for-index-reads | なし | SampleSheet.csvに記載されているインデックス配列に基づいてdumultiplexを実行する |
bcl2fastqによるFastqファイルの生成
仮想マシン内にbcl2fastqがインストールできたら、次はPart1でダウンロードしたRunデータフォルダをホストマシンの共有フォルダ(今回はホストマシン側にUbuntu_hsfとうフォルダを作成した)に入れます。
そうすることで、仮想マシン上で共有フォルダにあるRunデータを対象にbcl2fastqによる解析が可能となり、出力結果をホストマシンで取得することができます。
次にサンプルシートを加工していきます。[Data]行より下をすべて削除したのち、Edit_SampleSheet.csvという名前で保存します。これでbcl2fastqの機能では、分析に供したサンプルへdemultiplexされなくなりました。demultiplexは後に解析パイプラインClaidentを利用して行います。
次に仮想マシン側に作成していたUbuntu_gsfという共有フォルダ上で、右クリック、Open terminalをクリックしてTerminalを開き、以下のコマンドを実行します。
|
1 2 3 4 5 6 |
$ bcl2fastq --processing-threads 8 \ --use-bases-mask Y150n, I8, I8, Y150n \ --create-fastq-for-index-reads \ --runfolder-dir (Runのフォルダ名) \ --sample-sheet /(Runのフォルダ名)/(改変したサンプルシート名) \ --output-dir output |
※–use-bases-mask の値が間違っていたので修正しました。Y151n → Y150n
問題なく実行されれば、まずoutputフォルダが作成されて、その中に
- Undetermined_S0_L001_I1_001.fastq.gz
- Undetermined_S0_L001_I2_001.fastq.gz
- Undetermined_S0_L001_R1_001.fastq.gz
- Undetermined_S0_L001_R2_001.fastq.gz
の4つのfastq.gzファイルが生成されます。
タグ配列とそれに対応するサンプル名をサンプルシートに記載していれば、Reportsフォルダ内に配列を何リード配分したかなどの情報がhtml形式で記載されます。
生成されたI1とI2はインデックス配列、R1とR2はPaired-endで読んだ際のFoward側とReverse側それぞれの配列が割り振られています。下記図でいうとi5-SP1-Insert, i7-SP2-Insertのセットです。

ここまでこれば、Claidentを利用したdemultiplexや以降の解析の準備が完了です。
参考
- Development and evaluation of PCR primers for environmental DNA (eDNA) metabarcoding of Amphibia
(https://doi.org/10.1101/2021.10.29.466374) - DADA2 と Claident を用いた short-read amplicon sequence のデータ解析
(https://ushio-ecology-blog.blogspot.com/2019/11/20191129blogger0002_30.html) - Metabarcoding and DNA barcoding for Ecologists: Sequence analysis
(https://github.com/astanabe/MetabarcodingTextbook/blob/master/metabarcodingtextbook2.ja.pdf) - bcl2fastq2 Conversion Software v2.20
(https://jp.support.illumina.com/content/dam/illumina-support/documents/documentation/software_documentation/bcl2fastq/bcl2fastq2-v2-20-software-guide-15051736-03.pdf)
免責事項
十分注意は払っていますが、本記事の情報・内容について保証されるものではありません。本記事の利用や閲覧によって生じたいかなる損害について責任を負いません。また、本記事の情報は予告なく変更される場合がありますので、ご理解くださいますようお願いします。






