この記事の内容
pythonの環境について
pythonを使うことが多くなってきました。初心者にも取り組みやすいプログラミング言語だなと実感しながら使ってます。
pythonにはプロジェクトごとにPCのシステム環境とは独立した開発環境を構築することができます。
パッケージの依存関係を保ったり、ミスったときに環境を再構築したりする仮想環境という機能?管理法?のことです。
以前のPCでは綺麗に管理できてなかったことや、3月頭に新しいPCを購入して環境を再構築する必要が出てきたので、備忘録を残しておこうと思います。
仮想環境が必要だなと思った理由
使い始めは仮想環境を作成して使用していましたが、プロジェクトレベルで何かを進めるのではなく、小さいものがパラパラと出来上がる程度で、時折、仮想環境の中での作業を忘れることが多くなりました。
仮想環境外だと、パッケージをインストールしたり設定を変えたりした際には、システムの状態を変化させていることになります。その状態で面倒事が発生しすると、すべてを一度消してしまいたい気持ちを抑えながらトラブルシューティングする羽目になります。
常に仮想環境に入って作業をするような状態を保ち、扱いやすい管理システムを使うことが自分には合っているのではないかと思いました。
pythonとAnaconda
pythonを使っていくうえで、様々なパッケージを利用しますが、利用方法が2種類あります。
一つは、Python Package Index(PyPI)です。
誰でも自由に自分のソフトウェアを登録して公開できるようになっており、公開されているpythonソフトウェアのほとんどはPyPIから入手できます。
pipというコマンドでPyPIからソフトウェアを取得することができます。
もう一つは、データサイエンスに使用されることが多いツールやライブラリを包括的に提供しているAnacondaと呼ばれる実行環境で、conda
2通りの方法で、pythonのソフトウェアを入手することができますが、基本的に両者を共通して使うことは推奨されていないようです。
どちらの方法を使うかで、管理の仕方が変わるということになります。
Condaかpipどちらを使うのか
仮想環境が作成できるメジャーな開発プラットフォームを挙げてみると、
- Anaconda – conda
- miniconda – conda
- pyenv – pip
などが、調べてみるとよく見る代表格だと思います。機能面で見ると横並びではないですが、カテゴリとしてはこんな感じでしょう。
pyenvは一度導入してみたことがあるのですが、今どのような状態なのかが正直分かりにくく、私には合いませんでした。
Anacondaは一定の条件下での商業利用について有償化されるらしいので、今回はCondaベースのMinicondaで環境を構築していきたいと思います。
minicondaをインストールする
MinicondaはAnacondaの必要最低限パックみたいなものです。Anacondaはなんでも入っている一方でかなりの容量を占領します。自己管理という意味でもMinicondaを導入して、必要なものを都度入れていく方針がいいと思います。
wgetを使ってMinicondaをインストールするスクリプトをダウンロードします。以下のサイトで自身のpython3のバージョンにあったMinicondaのインストーラーのURLをコピーします。
https://docs.conda.io/en/latest/miniconda.html
python3.8.5、WSL + Ubuntu LTS 20.04で運用しているので、Miniconda3 MacOSX 64-bit bashをインストールしました。
|
1 2 |
# wgetを使ってMinicondaをインストールするスクリプトをダウンロード $ wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh |
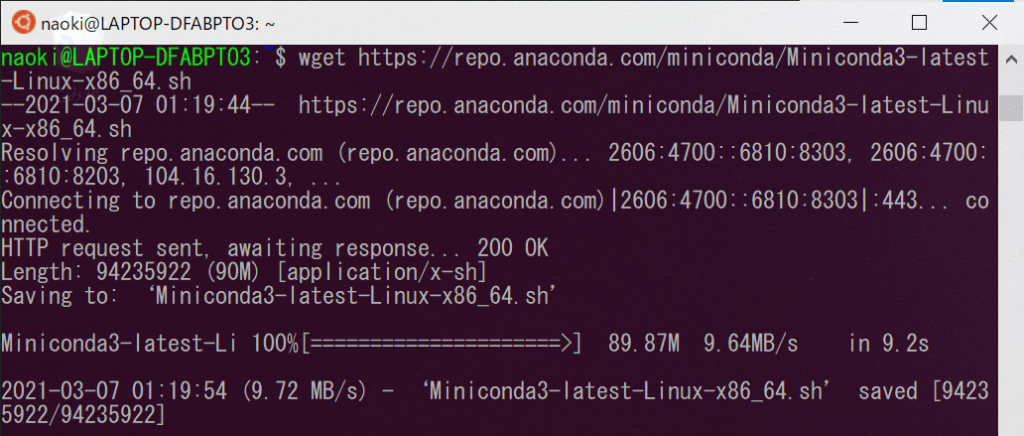
Miniconda3-latest-Linux-x86_64.shがダウンロードされました。
ダウンロードしたシェルスクリプトを使ってMinicondaをインストールします。
|
1 |
$ bash Miniconda3-latest-Linux-x86_64.sh |
インストール中にインストールする場所についてなど、いくつか質問が出てくるので、問題なければYesと答えておきましょう。
こんな感じで(base)~と頭についた状態ならOKです。デフォルトのConda環境であるbaseに入った状態です。
インストーラーはいらないので消しておきます。
|
1 |
$ rm Miniconda3-latest-Linux-x86_64.sh |
condaコマンドが使えるか試してみましょう。
|
1 |
$ conda |
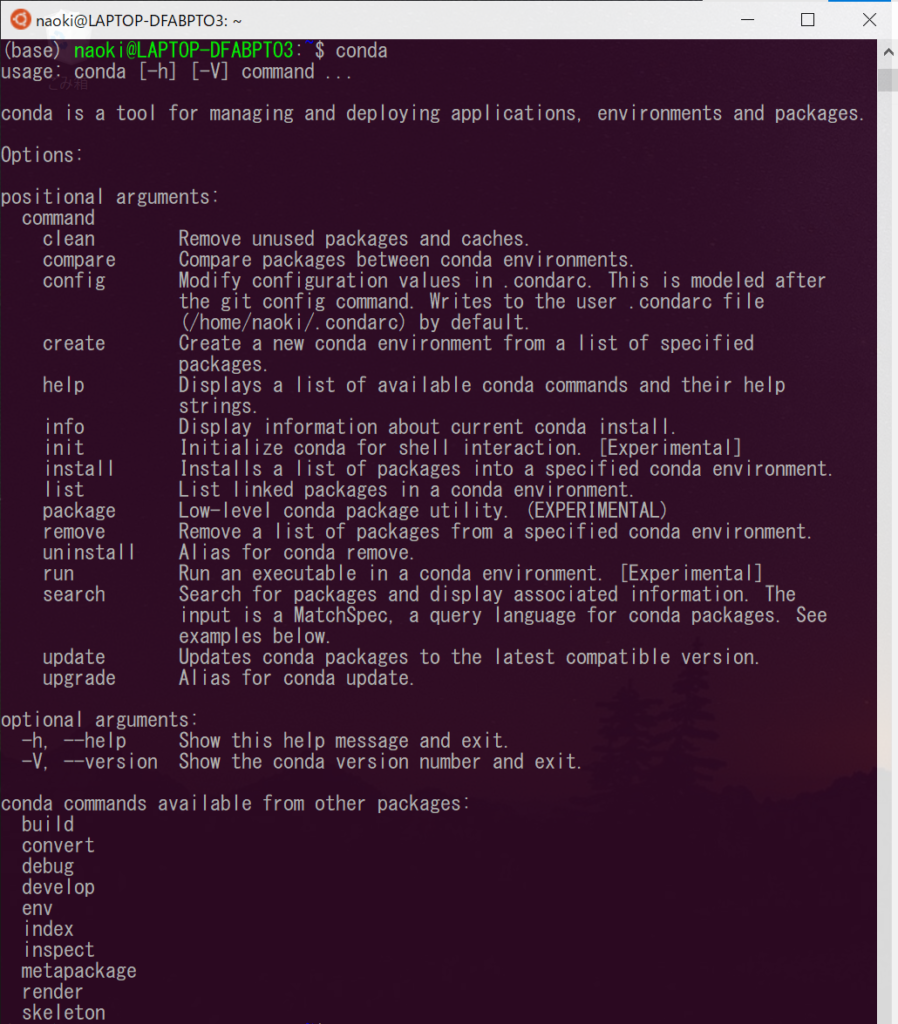
Miniconda導入時にどんなパッケージがダウンロードされたか確認してみましょう。
|
1 2 |
# インストールされたパッケージの確認 $ conda list |
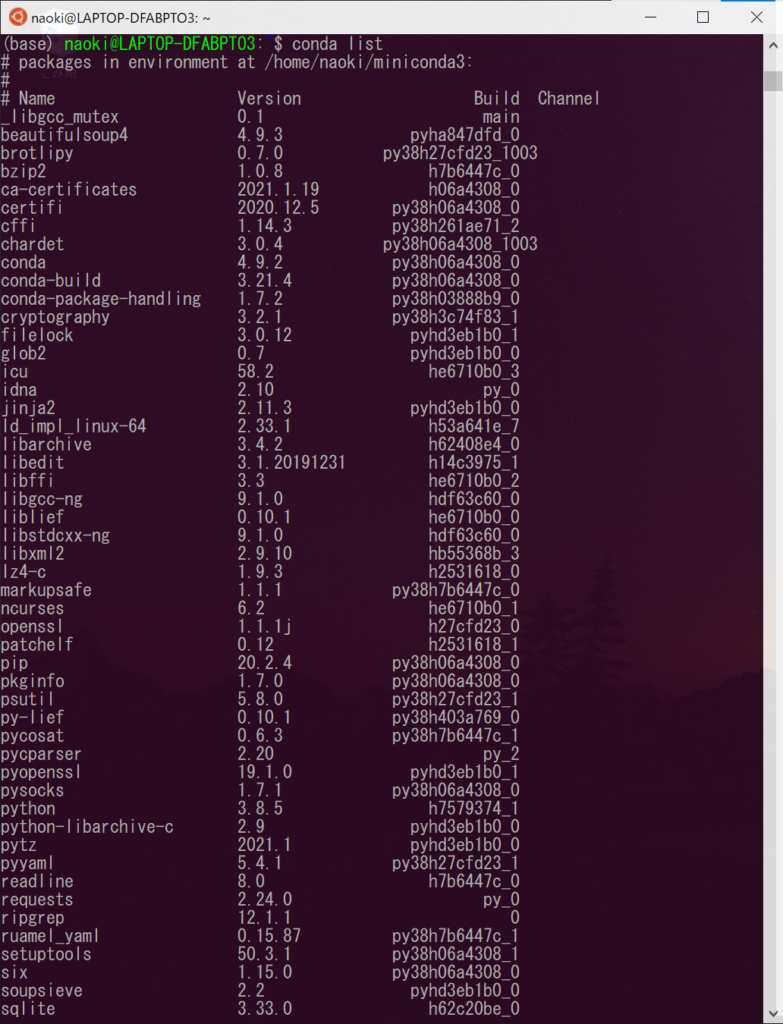
base環境から出てみましょう。
|
1 |
$ conda deactivate |
試しにpython3.9.1の仮想環境を作ってみましょう。
|
1 |
$ conda create --name py39 python==3.9.1 |
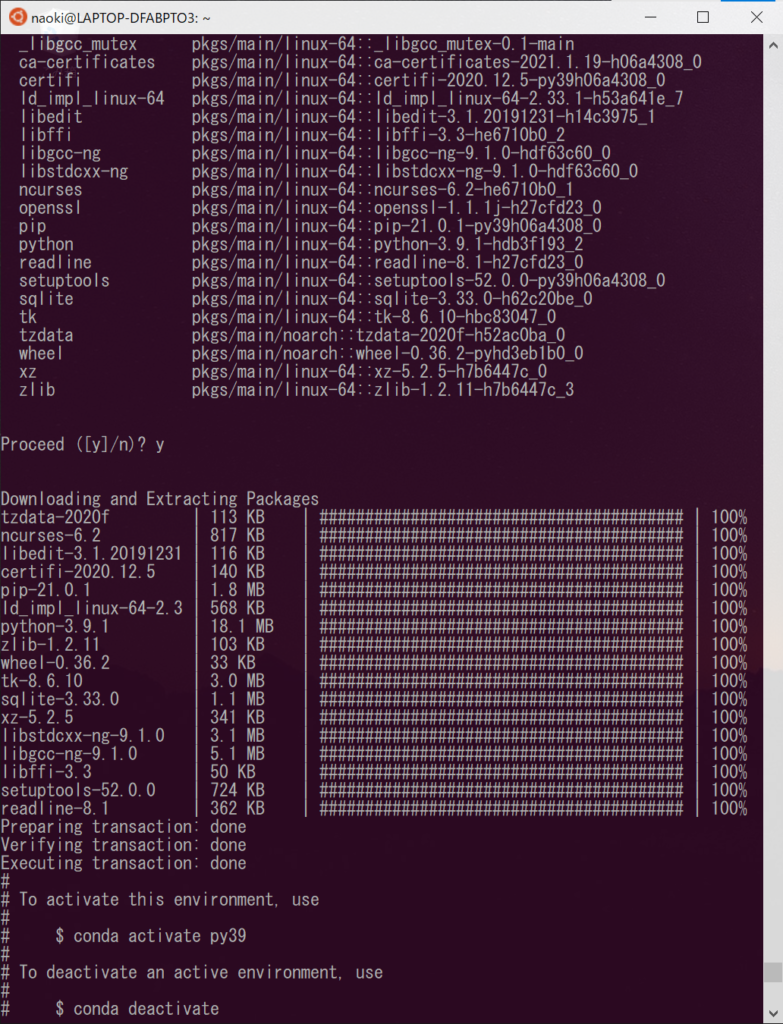
作成した仮想環境に入ってみます。
|
1 |
$ conda activate py39 |
頭に(py39)がつけばばっちりです。
作成した仮想環境一覧は以下のコマンドで確認できます。
|
1 |
$ conda info -e |
pythonのバージョンを確認してみます。
|
1 2 |
# Python 3.9.xと返ってくるはず $ python3 -V |
ここまで来たら、いくつかパッケージをインストールしてみましょう。
mambaがcondaより高速でいいと聞いたので導入してみたいと思います。 導入は以下の記事を参考にしています。
https://qiita.com/aical/items/4d6ab88ffcf312cb7590
開発者のGithubは以下になります。
https://github.com/mamba-org/mamba
|
1 2 3 |
# -cオプションはチャネルの指定 # conda-forgeというコミュニティを指定 $ conda install -c conda-forge mamba |
mambaがインストールできました。
使うようなものを一通り入れていきます。pythonソフトウェアはmambaを使ってインストール。
Blast+
python関係ないですが、ローカルのBLASTはよく使うので、コマンドでインストールします。
|
1 2 3 4 5 |
# aptコマンドでインストール $ sudo apt install ncbi-blast+ # 使えるか確認 $ blastn -h |
Biopython
NCBIからの配列取得には必要なので、biopythonをインストール。
|
1 |
$ mamba install biopython |
まとめ
- Minicondaをインストールしました
- condaベースで仮想環境を作成する方法を記載しました
- systemはpython3.8, 仮想環境の中ではpython3.9を使用できるようにしました
- Conda同様にpythonパッケージを管理できるmambaをインストールしました
- local環境でblastnが実行できるようにblastnをインストールしました
- バイオデータの解析を行う際に必要になるライブラリ集であるbiopythonをインストールしました
参考文献
- pythonとAnaconda
https://www.python.jp/install/docs/pypi_or_anaconda.html - condaの代わりに高速なmambaを使う
http://kazumaxneo.hatenablog.com/entry/2021/02/11/073000 - Mambaを使った高速condaパッケージ管理 (python)
https://qiita.com/aical/items/4d6ab88ffcf312cb7590 - はじめてコマンドを使う人向けの開設
http://kazumaxneo.hatenablog.com/entry/2019/10/16/122613 - Understanding Conda and Pip
https://www.anaconda.com/blog/understanding-conda-and-pip - Condaコマンド
https://www.python.jp/install/anaconda/conda.html - mamba-org
https://github.com/mamba-org/mamba
免責事項
十分注意は払っていますが、本記事の情報・内容について保証されるものではありません。本記事の利用や閲覧によって生じたいかなる損害について責任を負いません。また、本記事の情報は予告なく変更される場合がありますので、ご理解くださいますようお願いします。






