水生昆虫のDNAメタバーディングのための新規PCRプライマーセットの開発といくつかの隠蔽種の発見
Development of novel PCR primer sets for DNA metabarcoding of aquatic insects, and the discovery of some cryptic species
昆虫のDNAバーコーディングのためのユニバーサルプライマーの開発論文です。本論文のプライマーを環境DNAに適用できれば水棲昆虫の検出で課題の一つとされていた”解像度”が向上するかもしれません。
今回は技術的な部分を中心に訳しており、隠蔽種等に関する内容は一部省いております。
また、2022年1月現在は本稿はプレプリントなので、正式に発表される際には内容が変わっている可能性があるのでご注意ください。
※1/3にLimnologyから公開されておりました。(1/22)
DOI : https://doi.org/10.1007/s10201-022-00710-5
※日本の研究.com掲載プレスリリース(1/22)
URL : https://research-er.jp/articles/view/118359
こちらの方が論文をもとにした記事のため正確な可能性があります。
この記事の内容
Abstruct
DNAバーコーディングは標準的な遺伝子領域を用いて、迅速かつ正確に自動化された種の識別を可能にする強力なツールです。また、DNAバーコーディングは、生物学の様々な分野で、隠蔽種や希少種の存在を明らかにしたり、河川の生物相をモニタリングするなど、環境科学の分野で強力なツールとなります。
近年の生物多様性の減少は世界規模で最も深刻な環境問題の一つとなっています。
DNAバーコーディング技術においては、ユニバーサルプライマーセットの開発が必要です。この研究では、すべての昆虫グループのDNAバーコーディングのためのユニバーサルプライマーセットの開発を試みました。
その結果、ほぼすべての昆虫グループのDNAバーコーディング領域に対応したユニバーサルプライマーセット(保存的な領域の間に超可変領域を挟むように設計されたプライマーセット)だけでなく、データベースに登録するためのより長い配列に対応したプライマーセットの設計にも成功しました。
ミトコンドリアDNA16s rRNA領域でDNAバーコーディングを行った結果、14目43科68種について増幅に成功し、ミトコンドリアDNA 12s rRNA領域でDNAバーコーディングを行った結果13目42科66種について増幅に成功しました。
これらのプライマーセットで増幅されるDNAバーコーディング領域のDNA断片は、いずれも約200bpと短いことが大きな特徴で、より長い断片配列があれば、DNAデータベースへのデータ登録レベルが向上します。
このようなデータベースの充実は、生物多様性や遺伝子の多様性をより正確に評価するための強力なツールとなるでしょう。
Keywords: Biodiversity, DNA barcoding, eDNA, mtDNA mtDNA 12S rRNA, 16S rRNA
Introduction
昆虫は地球上で最大かつ最も多様なグループであり、約100万種が記載されています(Grimaldi and Engel 2005 ; Tojo etal.2017 ; Stork 2018)。
DNAバーコーディングは、標準化された遺伝子領域を使用して、迅速、正確、自動化可能な種の識別を提供するシステムです(Hebert and Gregory 2005)。
一般的に多くの昆虫は形態的特徴に基づいて識別されます。ですが、形態で種を識別するのは専門知識が必要となります。
このような状況においても、DNAバーコーディングは標準的な短いDNA断片を配列決定することで種を迅速に識別可能です(Hebert and Gregory 2005 ; Miya etal.2015)。また、形態分類に適さない幼虫の標本や不完全な状態の標本の種の識別にも効果を発揮します。
この方法は、迅速的でかつ再現性が高いため、幅広い種に使用して長期的なモニタリングを行い、生物多様性を理解することができます(Hänfling etal.2016 ; Uchida etal.2020 ; Chucholl etal.2021)。加えて、DNAバーコーディングは隠ぺい種や希少種の存在を特定する効果的なツールでもあります(Herbert et al.2004)。
DNAバーコーディングによる形態識別や在不在を推定するためには、DNAメタバーコーディング用のユニバーサルPCRプライマーセットを開発する必要があります。
代表的なものとしては、
- 魚類 MiFish : Miya et al.2015
- 哺乳類 MiMammal : Ushio et al.2017
- 甲殻類 (十脚目) MiDeca : Komai et al.2019
があります。
昆虫に関しては、ミトコンドリアDNAのチトクロームcオキシターゼサブユニット (通称COI)領域に設計された、Folmerのユニバーサルプライマーセットが頻繁に使用されます(Folmer et al.1994)。
しかし、COIはアミノ酸に翻訳されるタンパク質をコード領域であり塩基配列の保存性が高くない(Deagle et al.2014)。特に多型はコドンの3番目の塩基に集中する傾向があり、短いDNA断片を増幅するための汎用性の高いプライマーセットの開発には適さないと考えられる。
また、昆虫以外の環境DNAメタバーコーディングの研究では、リボソームrRNA領域内にユニバーサルプライマーが開発されています。
DNAバーコーディング領域のDNA断片の理想的な特徴は以下の通りです(Miya et al.2015, Valentini et al.2009)
- 特定の昆虫種を確実に同定するために、完全に同一か、同種の他の個体との配列差が少なく(種内多型が少ない)、他の種との配列差は明確である
- すべての昆虫を増幅できる相同な標準領域
- 対象領域が未記載種を容易に分類群(属・科)に割り当てることが可能な十分な系統情報を持つ
- 保存性と信頼性が高く、堅牢なDNA断片であること
- 短いDNA断片の増幅に適しており、昆虫の種を正しく特定するために十分な配列の多型を含んでいること
上記内容を踏まえて、まずこの研究で開発されたプライマーセットは、
- すべての昆虫(特に水棲昆虫)に適用可能
- 対象領域は約200bp
- 近縁な種でさえも、確実に区別することができる
といった特徴を有するように設計しました。
そのため、水環境中に存在する断片化された環境DNAに対しても有効性を示すと考えられます。
ただし、データベースの領域を参照するデータベースを充実化させるには、多大な労力と費用が掛かります。
そこで、より効率的にデータベースを充実させるために、DNAバーコーディング用の短い断片だけでなく、系統解析にも利用可能な目的のバーコーディング領域を含む長い断片も増幅する汎用性の高いユニバーサルプライマーセットを設計しました。
より長い断片も増幅できるこのプライマーセットは、系統研究に最適な手法として採用され、環境DNAの参照データベースの充実につながると考えられます。
また、対象となる水生昆虫では、近縁な種がニッチ分化することで、それぞれがさまざまな河川のとても狭い範囲の生息域に適応することが知られています。
DNAメタバーコーディング用に選んだDNA領域に、近縁種間でも種の判別に有効な多型が含まれているかどうか、また、そこから隠蔽種を容易に検出できるかどうかについても調べました。
Methods
リボソームRNA上のユニバーサルプライマーを設計領域を選択するために、多様性を高めるために、さまざまな昆虫グループの全体または部分的なミトコンドリアDNAの配列をGenBankからダウンロードしました。
対象分類群は以下の通りです。
| subclass | 和名 | Order | 和名 | Family | 和名 |
|---|---|---|---|---|---|
| Pterygota | 有翅亜綱 | Ephemeroptera | カゲロウ目 | ||
| Pterygota | 有翅亜綱 | Odonata | トンボ目 | ||
| Pterygota | 有翅亜綱 | Pleoptera | カワゲラ目 | ||
| Pterygota | 有翅亜綱 | Hemiptera | 半翅目(カメムシ目) | Belostomatidae | コオイムシ科 |
| Pterygota | 有翅亜綱 | Hemiptera | 半翅目(カメムシ目) | Nepidae | タイコウチ科 |
| Pterygota | 有翅亜綱 | Hemiptera | 半翅目(カメムシ目) | Gerridae | アメンボ科 |
| Pterygota | 有翅亜綱 | Hemiptera | 半翅目(カメムシ目) | Corixidae | ミズムシ科 |
| Pterygota | 有翅亜綱 | Hemiptera | 半翅目(カメムシ目) | Corydalidae | ヘビトンボ科 |
| Pterygota | 有翅亜綱 | Trichoptera | トビケラ目 | ||
| Pterygota | 有翅亜綱 | Coleoptera | コウチュウ目 | Dytiscidae | ゲンゴロウ科 |
| Pterygota | 有翅亜綱 | Coleoptera | コウチュウ目 | Gyrinidae | ミズスマシ科 |
| Pterygota | 有翅亜綱 | Coleoptera | コウチュウ目 | Lampyridae | ホタル科 |
| Pterygota | 有翅亜綱 | Coleoptera | コウチュウ目 | Dryopidae | ヤマモガシ科 |
| Pterygota | 有翅亜綱 | Diptera | ハエ目 | Simuliidae | ブユ科 |
| Pterygota | 有翅亜綱 | Diptera | ハエ目 | Culicidae | カ科 |
| Pterygota | 有翅亜綱 | Diptera | ハエ目 | Tipulidae | ガガンボ科 |
| Apterygota | 無翅亜綱 | Diplura | コムシ目 | ||
| Apterygota | 無翅亜綱 | Archaeognatha | イシノミ目 | ||
| Apterygota | 無翅亜綱 | Zygentoma | シミ目 |
最初は、水生昆虫のDNAバーコーディングに焦点を当てていたため、水生昆虫のシーケンスのみを参照していましたが、水生昆虫にはさまざまな昆虫群が含まれているため、その結果を使用して、ほぼすべての昆虫群に適用できる遺伝子領域を探索しました。
16S rRNA領域については、ダウンロードしたすべての昆虫グループのすべてのデータセットを使用して、非常に用途の広い領域を検索しました。
ただし、mtDNA 12SrRNA領域についてはすべての昆虫に対して単一のセットを設計することはできませんでした。
したがって、3つのグループのそれぞれについてmtDNA 12S rRNA領域を増幅するために、3つの特殊なプライマーセットを設計しています。
- 1)不完全変態
- 2)トビケラを除くホロメタボラ
- 3)トビケラ。
これらのプライマーは、Miya et al. 2015に記載されている各プライマーの両端に対して3 ‘末端の相補性だけでなく、5’末端の相補性の高い領域も含めるように注意をして設計しました。
Testing the versatility of the newly developed primers
この研究で設計されたプライマーの多様性を評価するために、信州大学のTojo labに保管されているさまざまな昆虫グループから抽出および精製されたゲノムDNAを使用してPCR増幅を行いました。
PCR条件は以下の通りです。
| 試薬 | n = 1 (uL) | 備考 |
| 10× ExTaq Buffer | 1 | |
| dNTP(25mM MgCl2) | 0.8 | |
| Ex Taq ポリメラーゼ(5U /uL) | 0.05 | |
| Forward Primer | 0.25 | |
| Reverse Primer | 0.25 | |
| Genomic DNA | 1 | |
| Total Volum | 10 | ミリQなどでメスアップ? |
mtDNA 16SrRNAおよび12SrRNA領域のDNAバーコーディング領域のPCRプロトコルは次のとおりです。
| Step | Temparature (℃) | Time(s) | Cycle |
| 初期変性 | 94 | 60 | 1 |
| 変性 | 94 | 60 | – |
| アニーリング | 50 | 30 | 30 |
| 伸長 | 74 | 60 | – |
| 最終伸長反応 | 74 | 180 | 1 |
PCR産物は、ExoSAP-IT Express (Thermo Fisher Scientific)にて精製し、精製されたDNA断片の配列決定は、Eurofins Genomicsに外注しました。
BigDyeターミネーターサイクルシーケンスキットv3.1(ABI)は、ABIシーケンサーでシーケンスする前に使用されました。配列データは日本DNAデータバンクに掲載。
すべての配列データは、MAFFT v7.222を使用してアライメントし、系統解析は、MEGA 7.0.26を使用した近隣結合(NJ)法によって実施しました。
(Evaluation of interspecific variations and phylogenetic analysisの項は訳しておりません)
Results
Design of versatile primer sets for DNA metabarcoding
mtDNA 16S rRNA領域のDNAバーコーディング領域を増幅するために、ほぼすべての昆虫グループに適用可能なプライマーセットMtInsects-16Sを設計。
また、系統解析に非常に有用なDNAバーコーディング領域を含むより長いDNA断片のPCR増幅に使用できるプライマーセットAQdb-16Sも設計。
このプライマーセットは、MtInsects-16Sの参照配列を登録する際役立つと思われます。
また、mtDNAの12S rRNA領域では,
- Hemimetabola(外翅類)
- Trichoptera(トビケラ目(毛翅目))を除くHolometabola(完全変態上目)
- Trichoptera(トビケラ目毛翅目)
上記、3つのグループごとに,DNAバーコーディング領域を増幅するプライマーセットを設計した。
下図は今回設計されたプライマーとプライミング領域の一致率を表しています。
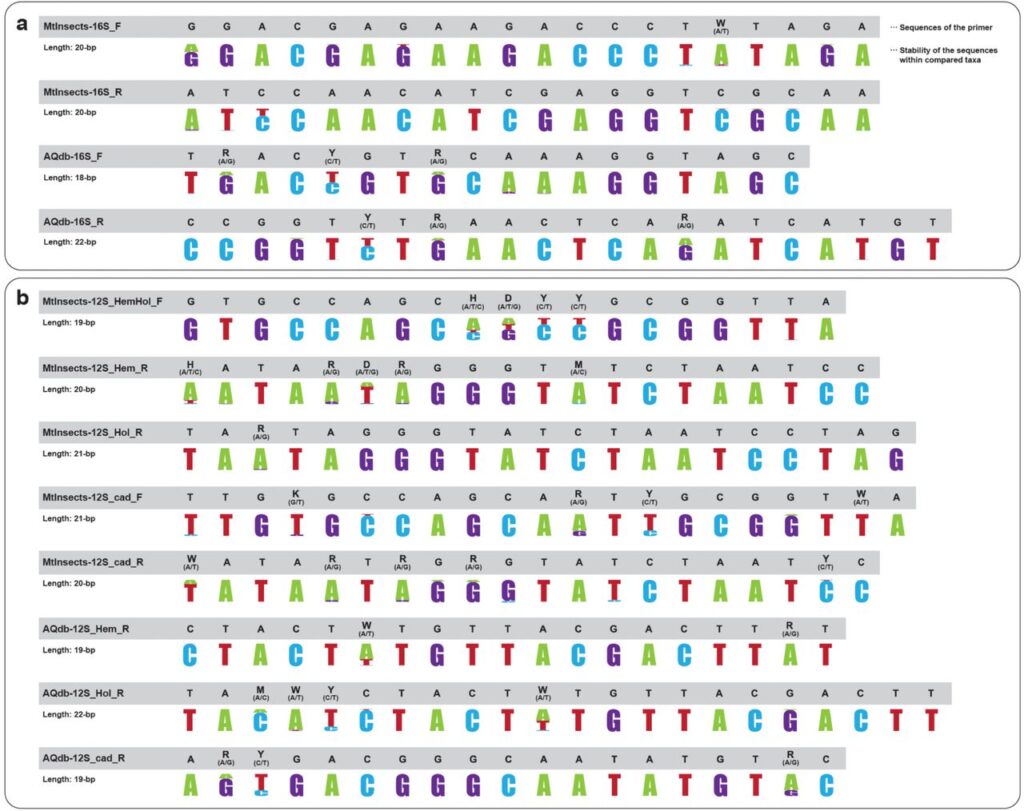
| Gene | Primer Name | Primer direction | Primer Sequence | Target group | Purpose |
| mtDNA 16S rRNA | MtInsects-16S_F | F | GGACGAGAAGACCCTWTAGA | All | DNA barcoding |
| MtInsects-16S_R | R | ATCCAACATCGAGGTCGCAA | All | DNA barcoding | |
| AQdb-16S_F | F | TRACYGTRCAAAGGTAGC | All | Pylogeny and database | |
| AQdb-16S_R | R | CCGGTYTRAACTCARATCATGT | All | Pylogeny and database | |
| mtDNA 12S rRNA | MtInsects-12S_HemHol_F | F | GTGCCAGCHDYYGCGGTTA | Hemimetabola & Holometabola | DNA barcoding & Pylogeny and database |
| MtInsects-12S_HemHol_R | R | HATARDRGGGTMTCTAATCC | Hemimetabola | DNA barcoding | |
| MtInsects-12S_Hol_R | R | TARTAGGGTATCTAATCCTAG | Hemimetabola | DNA barcoding | |
| MtInsects-12S_cad_F | F | TTGKGCCAGCARTYGCGGTWA | Trichoptera | DNA barcoding | |
| MtInsects-12S_cad_R | R | WATARTRGRGTATCTAATYC | Trichoptera | DNA barcoding | |
| AQdb-12S_Hem_R | R | CTACTWTGTTACGACTTRT | Hemimetabola | Pylogeny and database | |
| AQdb-12S_Hol_R | R | TAMWYCTACTWTGTTACGACTT | Hemimetabola | Pylogeny and database | |
| AQdb-12S_cad_R | R | ARYGACGGGCAATATGTRC | Trichoptera | Pylogeny and database |
Barcoding Primers
- Hemimetabola(外翅類)はMtInsects-12S_HemHol_FとMtInsects-12S_Hem_Rをバーコーディ
ング領域を増幅するためのプライマーセットとして使用した。 - Holometabola(完全変態上目)(Trichoptera(トビケラ目(毛翅目))を除く)についてはMtInsect-12S_HemHol_FとMtInsects-12S_Hol_Rをバーコーディング領域を増幅するためのプライマーセットとして使用した。
- Trichoptera(トビケラ目(毛翅目))はMtInsects-12S_cad_FとMtInsects-12S_cad_Rを使用してDNAバーコーディング領域を増幅した。
DataBase Primers
- Hemimetabola(外翅類)はMtInsects-12S_HemHol_FとAQdb-12S_Hem_R
- Holometabola(完全変態上目)(Trichoptera(トビケラ目(毛翅目))を除く)はMtInsects-12S_HemHol_FとAQdb-12S_Hol_R
- Trichoptera(トビケラ目(毛翅目))はMtInsects-12S_cad_FとAQdb-12S_cad_R
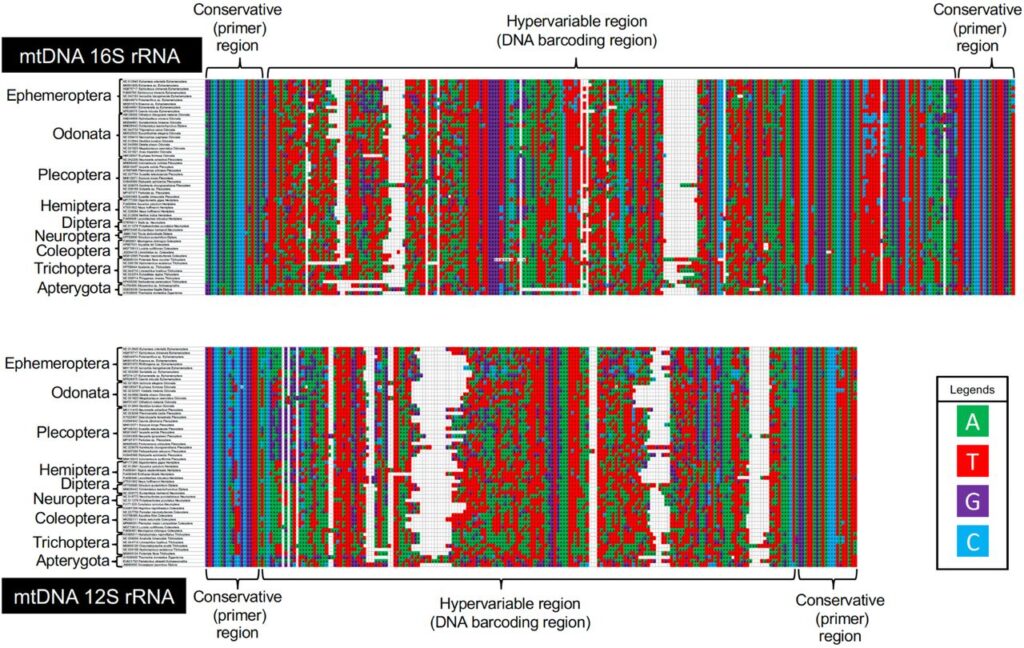
各プライマーの位置関係は図S2に示した通りです。
Versatility and nucleotide polymorphisms of the DNA
metabarcoding regions
16s rRNA領域に設計したDNAバーコーディングプライマーを使用して、14目, 43科, 68種、12s rRNA領域に設計したDNAバーコーディングプライマーを使用して、13目, 42科, 66種のDNAバーコーディングに成功したことを確認した。
注意点として、Tm値が約10℃異なる12s rRNA領域のプライマーセットを使用する場合には注意が必要です(GC配列が偏っていたため、Tm値を均一にできなかったため)。
また、本研究で開発されたプライマーセットがDNAバーコーディング領域で種を識別できるかどうか円形系統樹を使って確認しました。
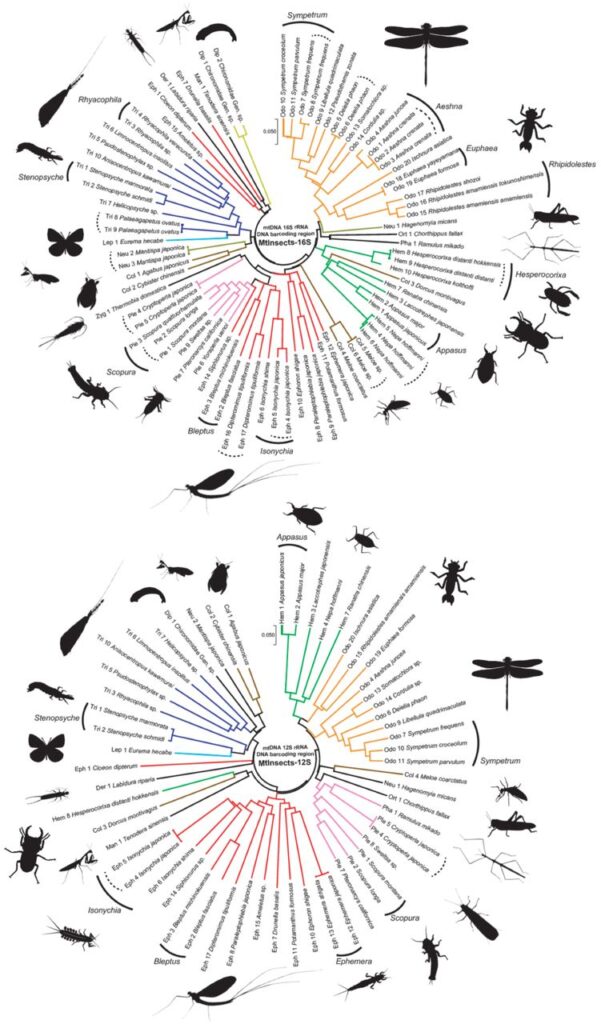
その結果、分析対象種においては配列を共有することはなく、同属の種においても各種を識別するのに十分な多型を有することが分かった。
ただ、2つの例外的なケースもありました。
まず、亜種関係にあるHesperocorixa distanti hokkensis(ホッケミズムシ)と Hesperocorixa distanti distanti(ミズムシ)は16s rRNA領域のプライマーで区別できていません。
これについては、COI領域でも区別不可であり、そもそも遺伝子マーカーでは区別ができないか、亜種レベルで実際に区別されていない可能性があります。
また、12s rRNA領域のプライマーにおいては、Ephemerajaponica(フタスジモンカゲロウ)とEphemera strigata(モンカゲロウ)が区別できませんでした。
これらは、日本で同所的に広く分布する姉妹種(sister species)である。種分化後に2種が再接触した地域では、種間の遺伝子移入?が確認されています(Takenaka et al. Unpub)。
そのため、多様性検出には大きな問題はないと考えています。
DNA barcoding method sufficiently sensitive to detect cryptic species
カゲロウ目の種であるEpeorus aesculusには、隠ぺい種が存在することが知られている(Ogitani and Nakamura 2008, Tojo 2010)。
本研究で開発した16Sと12Sの系統解析用プライマーを用いて、タイプ産地を含む日本アルプスの複数河川より採取されたEpeorus aesculusのDNAを増幅し、系統解析を行った結果、タイプ産地とは異なる分岐群(Clade B)が検出された(Fig.4)。
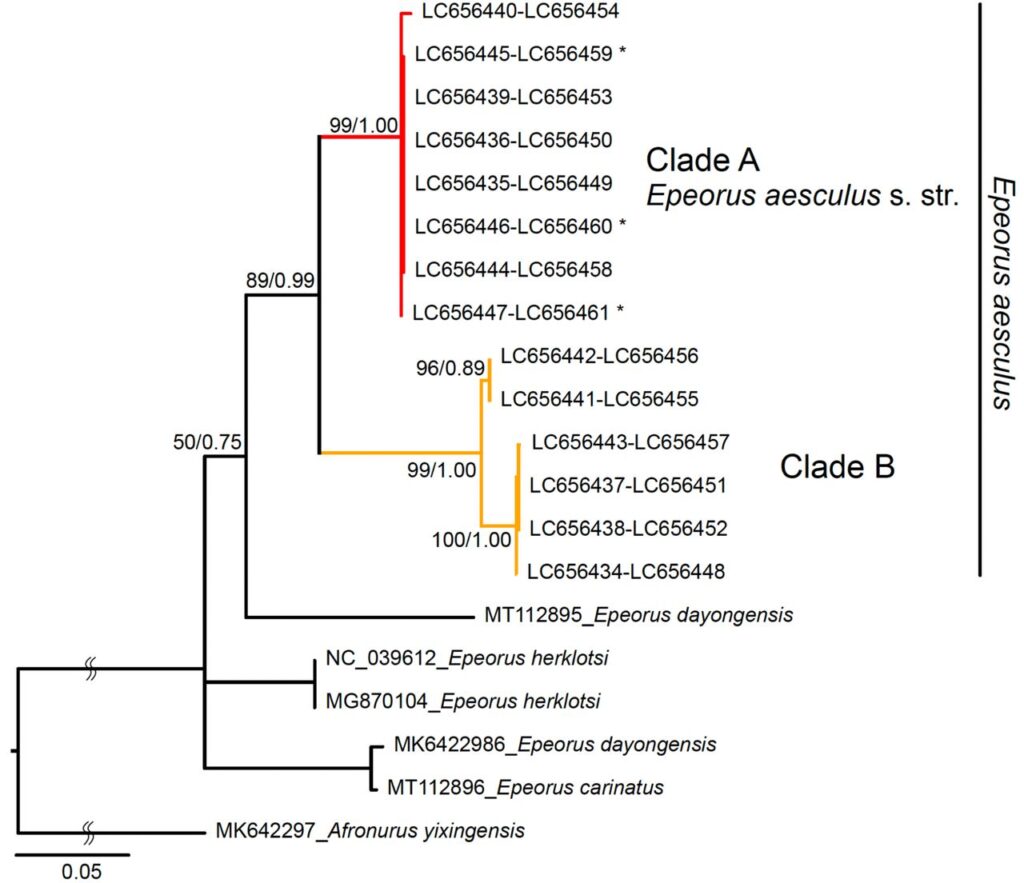
これは、1種として扱われているE. aesculusが遺伝的に分化し、隠ぺい種を有していることを意味しており、より詳細に調査を必要性があることを示しています。
Conclusion
この研究では、ユニバーサルプライマーセットと新たに選択された最適なDNAバーコーディング領域の開発に成功しました。
重要な特徴としては、増幅配列長が両プライマーとも約200bpと短いことです。
ほぼすべての昆虫グループに適用できるDNAバーコーディングまたはDNAメタバーコーディング用のプライマーセットを開発することにより、昆虫の種構成と種多様性を比較的簡単に長期的に監視できるようになります。
さらに、見落とされているか、形態学的に特定することが困難な多くの未記載の種および/または隠蔽種の発見を容易にすることも期待できます。
この研究では、ミトコンドリアDNAのリボソーム領域上てバーコーディング領域を確立しました。
ただし、リボソーム領域を使用して識別できなかった密接に関連する種は、将来的に識別可能になる可能性があります。
魚類のメタバーコーディング用プライマーである「MiFish」は、一部のグループの種を区別したり、バーコード領域を増幅したりできないことが報告されているため、特定のグループに焦点を当てた新しいプライマーセットが設計されました (Anguilla属:Takeuchi et al.2019 ;サケ科: Morita etal.2019 ; シクリッド科:Doble et al.2019)。
昆虫の場合、本研究で開発したリボソームDNA領域で種を特定できない場合、従来の標準的なDNAバーコーディング領域であるCOI領域で種を特定できる可能性があります。
このような場合は、既存のデータベースを有効に活用するのが最善です。
また、種の識別の精度と信頼性を向上させるために、複数の遺伝子領域を使用することをお勧めします。
最後に、DNAバーコーディング領域マーカーだけでなく、データベースに登録するためのより長いフラグメント配列用のプライマーセットも設計することができました。
より長いフラグメントを増幅できるこれらのプライマーセットは、系統解析にも使用できます。これにより、DNAデータベースへのデータ登録のレベルが向上します。
おわり






