魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish : マイフィッシュ)の開発者本人によるレビュー論文の和訳第二弾です。まだ、数パート続きます。研究課題を考える学生さんや新しく始める人などの仕事や研究の手助けになれば嬉しいです@しばた
MiFishメタバーコーディング:環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ
MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
目次
- Introduction
- 競合するUniversal PCRプライマーとの性能比較
- MiFishプライマーの分類学的最適化 (今回はここ)
- 望ましい実験室設定と改訂された実験プロトコルについて
- 新しいバイオインフォマティクスパイプラインと参照データベース
- さまざまな水生環境での実証研究のレビュー
- 海の魚類群集
- 淡水の魚類群集
- 河口域の魚類群集
- MiFishプライマーを用いた新しい技術の開発
- 他の生物へのMiFishメタバーコーディングの応用
- バルクDNAサンプルへのMiFishメタバーコーディングの応用
- さいごに
- 感想
1,2章の内容はこちら
3. MiFishプライマーの分類学的最適化
MiFishプライマーが他のプライマーより優れている点が多いことは実証されてきました。
その一方で、克服すべき点がいくつかあります。ここでは、”PCRのドロップアウトによる偽陰性”と”種判別領域が複数種間で一致する問題”の2点について研究例に沿って説明していきます。
PCRのドロップアウトによる偽陰性について
MiFish-E -E2
Miya et al.,2015ではPCR時のドロップアウトについて、水槽内で飼育されているサメやエイなどの軟骨魚類や板鰓類(elasmobranchs)がMiFishのオリジナルプライマーで増幅されませんでした。
著者らはプライマーと鋳型となるDNAのミスマッチに起因するPCRのバイアスに起因するのではないかとの疑念から、軟骨魚類や板鰓類(Elasmobranchs : E)のプライマーが引っ付く部位の配列変異に対応するために新しいプライマーペア(MiFish-E)を設計しました。
その後、このプライマーを使うことで、水槽内に飼育されている17種の硬骨魚類と板鰓類のすべてを検出することに成功しました。
しかし、その後の研究により、MiFish-Eではアニーリング温度の上昇とともに軟骨魚類や板鰓類の検出確率が著しく低下することが明らかになりました(Fig.6 From Doi et al.2019)。 そこで、フォワードプライマーとリバースプライマーの両方の5´末端に1塩基を追加したMiFish-E2を作成してプライマーの融解温度を上昇させることで、検出率の温度依存性を緩和しました(Miya and Sado 2019a)。
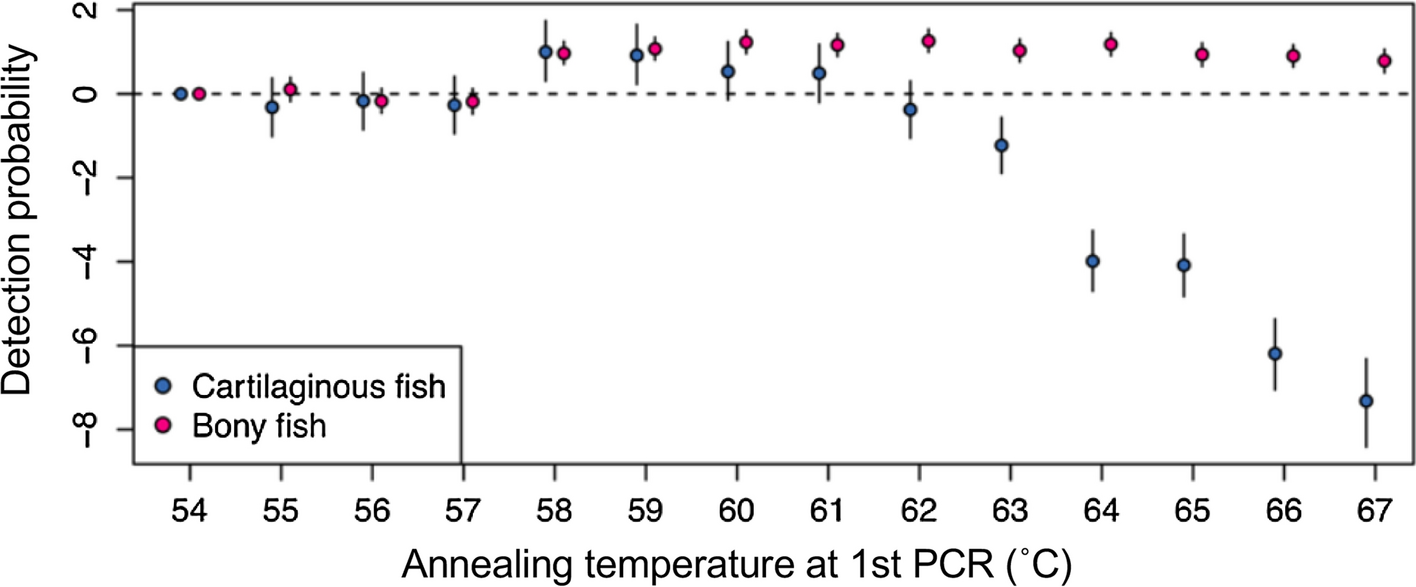
検出確率は多種占有モデル(Multispecies occupancy modeling)によって推定された。軟骨魚類の検出確率は62℃以上で減少している一方、硬骨魚類は58℃以上で安定していることが分かる
。Error barは95%信頼区間を示す。
MiFish-U2
日本中部の太平洋沿岸で一般的な岩礁に生息する海のカサゴ4属10種 : Pseudoblennius, Furcina, Ocynectes, Vellitor (Scorpaeniformes: Cottidae) は、検出されてもごくわずか、もしくは完全に検出されないことがありました。

MiFishのプライマーがひっつく部位をサンガーシークエンシング(DNA配列の決定)したところ、MiFish-Uのプライマーと鋳型DNAにミスマッチが1~6個あることが明らかになりました(論文Fig.7a)。
これらの10種の配列の変異に対応するように(MiFish-U2)を開発し、その後の野外調査により、地域の魚類群集組成をより正確に反映する合理的な結果が得られました(M. Miya , unpublish data)。
著者の研究室では、MiFish-U-F/R: MiFish-E-F/R : MiFish-U2F/R = 2 : 1 : 1のプライマーミックスを用いて、1st PCRを行い、海産魚類相からの環境DNAを増幅してきました(Miya and Sado 2019a)。
これら2つの例は、実際の調査を開始する前に、対象となる調査海域からのいくつかの水サンプルに基づいて、MiFishプライマーを用いた予備実験を行うことが重要であることを強く示唆しています。
予備実験の結果を捕獲による直接観察や水中で目視観察調査、文献調査による過去の生息記録と比較することで、予備実験の結果に欠落している一般的な種の中で明らかな偽陰性種(または種のグループ)を本調査の前に見つけることができます。
プライマーバイアスがかかりそうなその他の種について
日本の淡水魚類においてはヤツメウナギ(Lethenteron and Lampetra)やその他数種(アユ: Plecoglossus altivelis, ワカサギ属: Hypomesus spp.)が、MiFishメタバーコーディングのデータでは既知の分布や個体数とに比べて、過小評価されている可能性があり、それはやはりプライマーと鋳型DNAのミスマッチによるものと考えられています(論文Fig.7b, c)。
さらに類似した例がオーストラリアのパースでタツノオトシゴ類( Syngnathidae科)について報告されています。Nester et al. 2020では、新規開発されたプライマーによってタツノオトシゴ属を検出することができたものの、MiFishを含む既存のプライマーセットでは偽陰性でした。しかしこの知見は、MiFishのプライマーを用いて2種のタツノオトシゴを検出したTang et al. 2018とは対照的な結果でした。
種判別領域が複数種間で一致する問題
2属のマグロについて
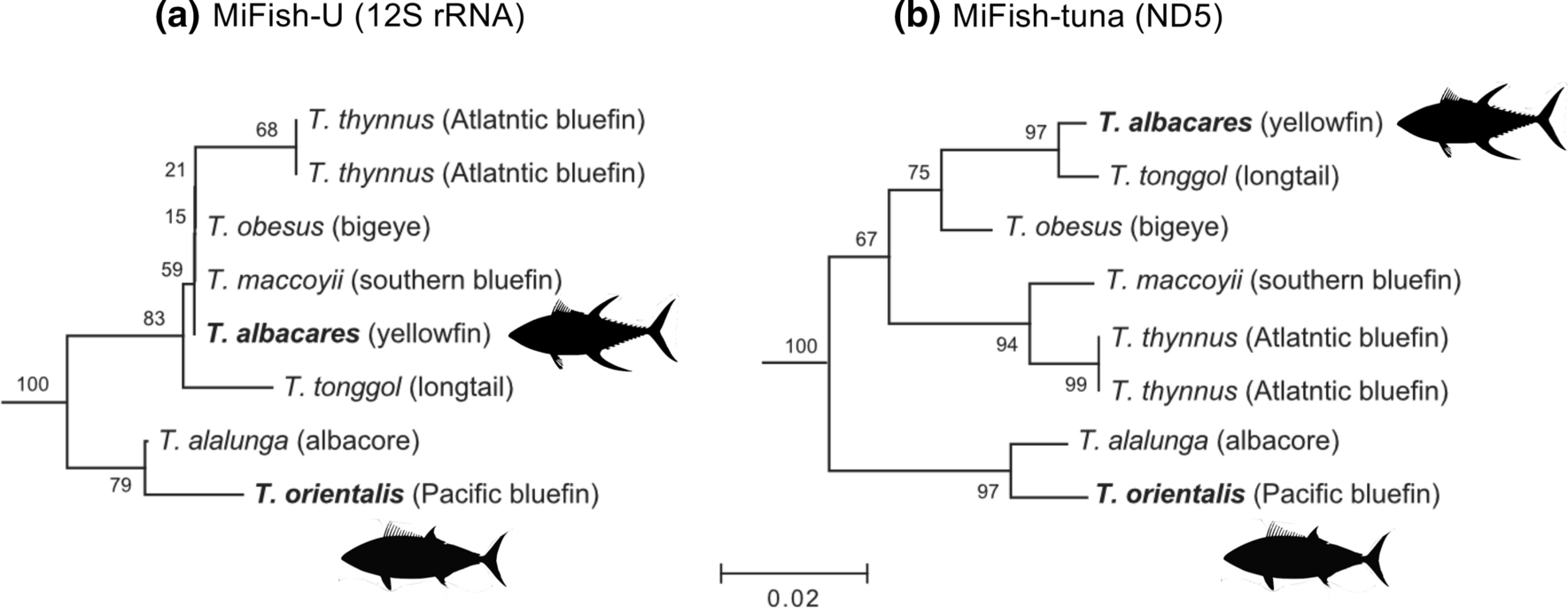
Thunnus属とScombridae属のマグロ7種はMiFish領域のDNA配列がそれぞれの種を見分けることのできる変異がないことを発見しました(論文 Fig.8a)。
7種のマグロから得たミトコンドリアDNAの塩基配列を目視で確認した結果、構成種間で十分な種間変異を持つND5遺伝子の一部(180bp)を増幅するための属に特異的なプライマー(MiFish-tuna-F/R)を設計し、MiFishメタバーコーディングによって正しく種が分けられることを確認しました。
領域の異なる12SとND5に設計されたMiFishプライマーを使ったマルチプレックスPCRのように、異なる領域を使ったプライマーの場合、それぞれのプライマーの増幅領域長を揃えることがシーケンス成功の為には重要です。
ウナギ属について
ウナギ属でも同様に種間でDNA配列の変異の欠如が確認されました。Takeuchi et al.2019では、ウナギ属16種のミトコンドリアDNA上のATP6遺伝子において、種間で5~22塩基、3亜種間では1~3塩基の差を持つ可変領域を増幅する新規プライマーセット: MiEelを設計しました。
MiFish-UとMiEelを使ったメタバーコーディングでは、ニホンウナギ(Anguilla japonica)の検出に成功しました。
また、Takeuchi et al. 2019はMiEelメタバーコーディングが複数のウナギ属が発生すると予想される産卵場の調査に有効であると主張しています。
実際、インドネシア諸島内とその周辺では8種のウナギ属の種が生息しており、6種が南太平洋西部で産卵することが知られています。
サケ科の種など
同様の例はサケ科のいくつかの種でも確認されました。Morita et al.2019では、ミトコンドリアDNAのND2領域に新規プライマーを作成しました。
シクリッド科やメバル属、トラフグ属などのミトコンドリアのコード領域において、種間での差が極めて少ない種群は環境DNAメタバーコーディングによる正しい生物種の割り当てのための代替的な遺伝子マーカーを見つける必要があります。
タンガーニーカ湖に生息するシクリッドにおいては、ミトコンドリア D-loop領域の超可変領域を使った新しいプライマーペアが開発されています(Doble et al.2019)。
第二和訳おわり
References
- Doble et al.2019
Testing the performance of environmental DNA metabarcoding for surveying highly diverse tropical fish communities: a case study from Lake Tanganyika. - Doi et al.2019
Evaluation of detection probabilities at the water-filtering and initial PCR steps in environmental DNA metabarcoding using a multispecies site occupancy model. - Miya et al.2015
MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species. - Miya and Sado 2019a
Multiple species detection using MiFish primers. In: eDNA Method Standardization Committee (ed) Environmental DNA Sampling and Experiment Manual Version 2.1. - Morita et al.2019
Ongoing localized extinctions of stream-dwelling white-spotted charr populations in small dammed-off habitats of Hokkaido Island, Japan. - Nester et al.2020
Development and evaluation of fish eDNA metabarcoding assays facilitate the detection of cryptic seahorse taxa (family: Syngnathidae). - Takeuchi et al.2019
New PCR primers for metabarcoding environmental DNA from freshwater eels, genus Anguilla.