MiFish: 魚類の環境DNAをメタバーコーディングするためのユニバーサルPCRプライマーセット:230を超える亜熱帯性海洋種の検出
魚類の環境DNA(eDNA)をメタバーコーディングするためのユニバーサルプライマーセット(MiFish-U/E : マイフィッシュ)の開発論文です。
880種のミトコンドリアのコンプリートゲノム配列と160種の板鰓亜綱(サメやエイ)のミトコンドリアの部分配列をもとに12s rRNA遺伝子の長可変領域(163bp-185bp)を標的として設計をしています。
このプライマーは魚類の種組成が分かっている沖縄県美ら海水族館の4つの水槽(a-d)でを使い、採水、デュアルインデックスのライブラリー調整と次世代シークエンサー(Miseq)によるハイスループットなペアエンドシーケンスで複数サンプル中に含まれる魚類の環境DNA配列を解読して検証を行っています。自然生態系での検証は水族館付近の岩礁帯(e-f)でしています。

水槽内の実験ではカスタムデータベースに登録されている海産魚類180種のうち59科123属にわたる168種(93.3%)を検出、野外調査では2L×4つのサンプルから93種が検出されています。
そのうちの63種は水族館の4水槽からは検出されておらず、論文全体を通して234種(70科152属)を検出されたことになります。検出された種組成を見るとサメやエイより高位の硬骨魚に至るまで分類学的に多様でかつ、沿岸から深海に生息する遠洋種および底生種など生態系においても非常に多様な種が挙がっています。
ここではメタバーコーディングによるアプローチが一般的な調査手法と比較して、非侵襲的、高効率、高い費用対効果、高感度であり、より大きな空間的・時間的スケールにおける魚類の資源管理と生態学研究に革命をもたらす生物多様性モニタリングの代替的(または補完的)ツールとして役立つ可能性を秘めている。と結論づけています。
DNAバーコーディングや分子生物学を専攻した方であれば周知の技術になっていますが、デュアルインデックスのライブラリー調整というのは2段階のPCRによって標的のDNA領域を増幅、NGSで解析する前に標的のDNAにサンプルタグ配列を付加後、分析するサンプルを混ぜ合わせる工程を指します(下図)。
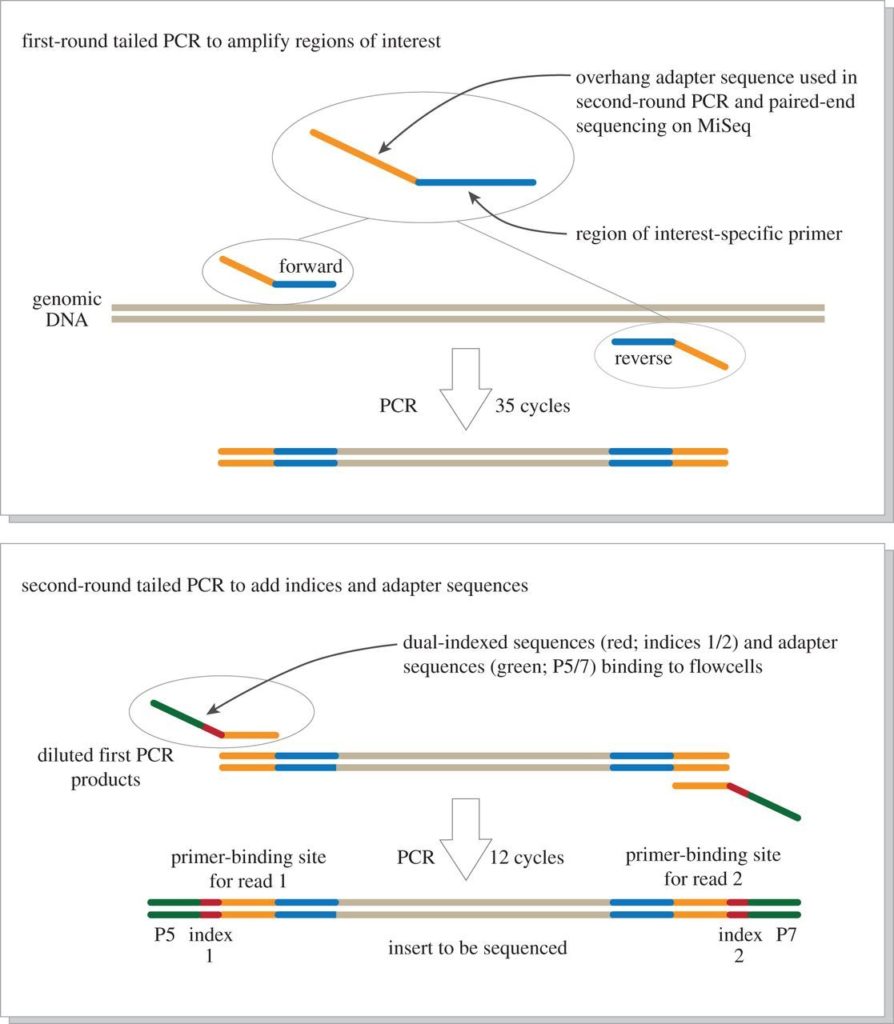
それぞれ、1stPCRプライマー、2ndPCRプライマーを使用します。一般的に耳にするMiFish-U/E/U2などは魚類におおよそ特異的(青色の部分)に作成されており、主な目的は様々な生物のDNAが含まれているサンプル内から魚類のDNAを増幅させることが目的で、1stPCRで使用します。
なぜ3つもあるかと言うと、魚類という種類の多い分類群を対象にすると、プライマーを作成した領域の一部の種においてはDNAの変異が入っている場合があります。DNA配列の変異があると増幅されない事があるので、そこをカバーするためです。また、このようなパターンの場合、プライマーを合成する際に決まった記号で複数の塩基を指定する混合塩基を用いる場合もあります。
そして、1段階目のPCRで魚類のDNAを数百万倍に増幅させた後、2段階目のPCRでそれぞれのDNAを混ぜ合わせても区別できるようにインデックス(標識 : 6塩基の配列パターン)となるDNA配列(赤色の部分)が含まれたプライマーを付加、長さの確認、濃度の調整で目的の濃度に調整したらNGSで配列を決定します。
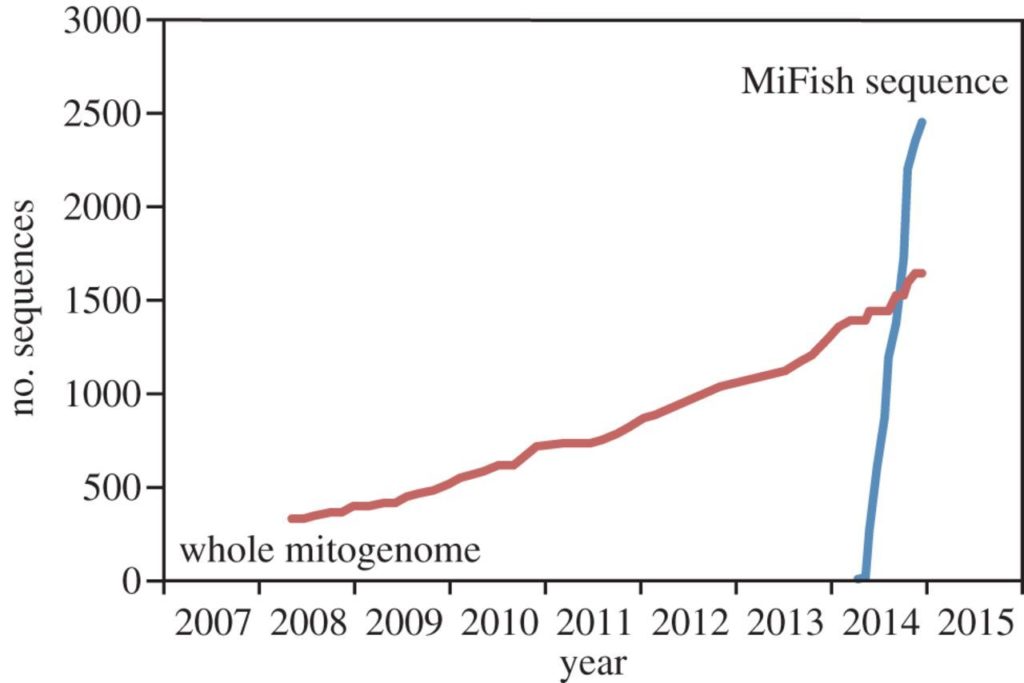
魚類を対象とした環境DNAメタバーコーディングの先行研究(Thomsen et al.2012, Kelly et al.2014 etc…)はいくつかありますが、上図のように日本での利用とデータベースの充実度を加味すると、千葉博の宮先生が開発したMiFishプライマーが最も実用性があると思います。
ただ、MiFishを用いたメタバーコーディングを行っていると、この魚類もMiFishは増幅が苦手なのかな?と考えられる種がいくつかおり、そういった種に対しては改良プライマーを作成してやるのが良いと思います。
また1回の分析での網羅性などを加味すると、この手法は生物多様性モニタリング≒生物相調査の代替的または”補完的”ツールとして利用するのが良いと現状は思いますが、使い方によっては一般的な認識よりもポテンシャルを発揮できるそんなツールです。
常に補完的な手法であることを前提にしつつも、技術の向上により網羅性の高い手法になるよう研究者だけでなく私たちユーザー側も情報共有や工夫を凝らしていきたいですね。@しばた
画像はジャーナルのCC BYに従う形で、記事で紹介したオープンアクセスの論文中の画像または、自身で作成したものを使用しています。






