採水の仕方は皆さんとても迷うところだと思います。やっぱり野外で同じ環境はないので、方法を一般化するのはとても難しいです。
なので、論文などで検証されているものを参考にして、可能な限り間違っていない方法でアプローチしていくのが良いと思います。もし、より良いアイディアがあるなら研究者とどんどん検証していきましょう。
今回は魚類を対象とした場合に、採水の仕方で検出される魚類相がどのように異なるのかについて検証した論文を解説します。また、自身の知識の整理と考えをまとめるような書き方をしています。@しばた
淡水魚類群集の環境DNAメタバーコーディングにおけるサンプルプーリングの有用性と限界
Ficetraが2008年に大型脊椎動物を対象とした環境DNAを発表してから約10年近く経った2017年でも環境DNAのサンプリングデザインの開発はまだ確立されていない状態です。
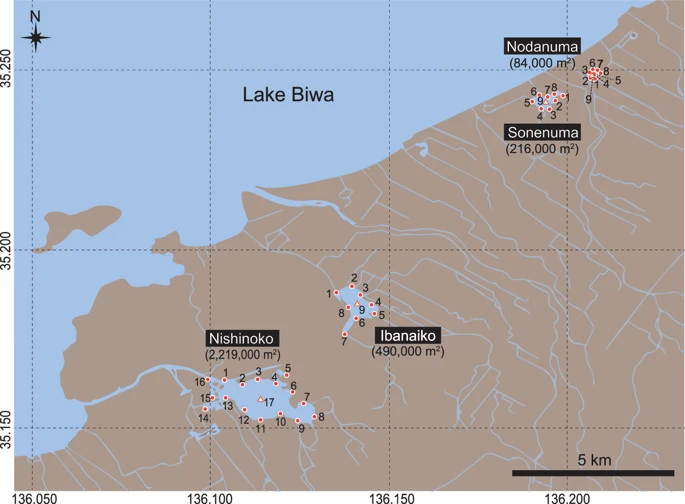
この研究では、MiFishを使った環境DNAメタバーコーディングにおいて、琵琶湖周辺に点在する4つの内湖(野田沼, 曽根沼, 伊庭内湖, 西の湖)の沿岸の複数箇所で採水して、個々のサンプルをそれぞれで分析する場合と、1つにプールして分析する場合の魚類の種組成を比較しました。
メソッド
採水
・野田沼:65m, 曽根沼:130m, 伊庭内湖:230m,西の湖:580m 間隔
・個別サンプル:各地点500mL
・プールサンプル:同じ地点で250mLずつ採水してまとめる
・ろ過はGF/Fフィルター(0.7μm)を使って現地ろ過
抽出
・Yamanaka et al.2016のようなスピンカラムを使った抽出法
・精製はDNeasy Blood and tissue kit
・溶出量は100μL
解析
・現行のweb MiFish pipelineと同様の工程で解析
・魚類群集の解析はRのveganやpheatmapを使用
結果と考察
個別に分析した場合、野田沼:31, 曽根沼:22, 伊庭内湖:33, 西の湖:31種の魚類が検出された一方、サンプル間で中程度の空間自己相関が検出されました。
その一方、それぞれの湖ごとにサンプルをプールして分析した場合、1st PCRの繰り返し数を15回に設定すると野田沼:30, 曽根沼:20, 伊庭内湖:29, 西の湖:27種が検出されました。
※空間自己相関とは、一言でいうと、近しいものは似ていて遠いものは異なる性質のことです。
ある地点で魚採りをした場合に50cm離れた所と10m離れた所では、採れた魚の種構成は50cm横より10m横の方が違ってくるはずです。環境と生物の種組成の違いなど、比較研究における調査設計において、空間自己相関は常に気を付けておく必要があると言えます。
下図は種数累積曲線と呼ばれる図です。
サンプリングによる種の増加をランダム化して累積することで、調査地点に生息する種数を推定するします。
縦軸が種数で横軸が個別サンプル(Individual)はサンプル数をプールサンプル(Pooled)の場合は1st PCRの繰り返し数を示しています。
青・赤色の影の部分は95%信頼区間です。
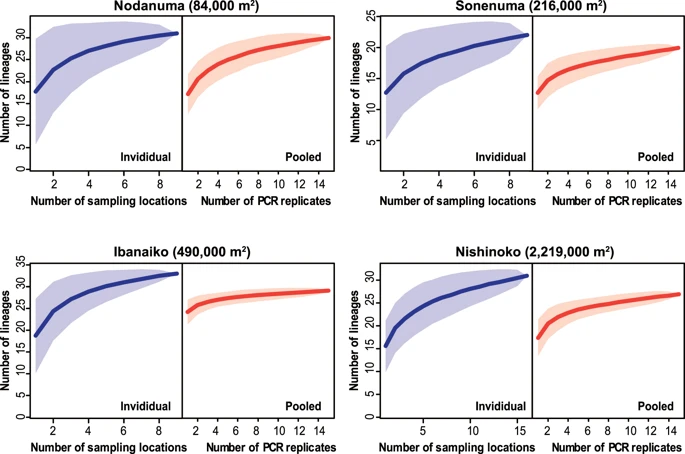
一つの目安として、累積曲線が定常状態に近い形を示していれば、今回のサンプリングで検出できるであろう種数をおおよそ網羅できていると言えます。
野田沼(左上)などは両サンプリング法とも定常状態に近いので、さらにサンプリング数を増やしても検出される種数の増加は見込めないだろうという風にとらえられます。伊庭内湖(左下)はプールサンプリングの累積曲線の形状が定常状態に近いのに対して、個別サンプリングはまだまだ増加しそうです。多分この差は、方法間の検出能力を表しているのではないかと考えられます。
影の部分のブレ幅の大きさも、どの湖でも個別>プールとなっているので、個別サンプルの方が1サンプル当たりの検出数が多い場合もあれば、少ない場合もある。各地点のサンプルをプールして1st PCRの繰り返しを設ける方が検出される種数と構成が安定的であると言えるかと思います。
また、1st PCRはよく4繰り返しで実施しているところをよく見ます。なのでプールサンプルの4繰り返しで推定される検出数は、個別サンプルをおおよそ何サンプルとるのと同等かというのを表してみました。

当然、個別サンプルは1サンプル当たりの検出種数にブレがあるので単純比較とは言えませんが、今回の場合だと、プールサンプルの1st PCR 4繰り返しは個別サンプリングを2サンプル以上分析しないといけないという風に見受けられます。また、西の湖のようにかなり大きな湖だと、複数地点を1つにまとめて分析するほうが、最低限のコストで、ある程度の種数を検出することが出来ると言えます。
ですが、それでもまだ多くの種の検出が望める西の湖の全個別サンプルで検出された種数の4/5種程度なので、プールサンプルの1st PCR 4繰り返しで調査地の種構成を反映できるかというと難しいでしょう。
環境DNAでされた種を可能な限り検出し、さらに捕獲されていない種を検出することを目指すのであれば、複数地点のサンプルを個別に分析するのがやはり良いと思います。
サンプリング手法間の特徴として、個別サンプリングで検出された総read数が<0.05%より小さい種は、プールサンプルからほとんど検出されませんでした。つまり、DNA量が少ない稀な種はプールサンプルからは検出されにくい可能性があります。
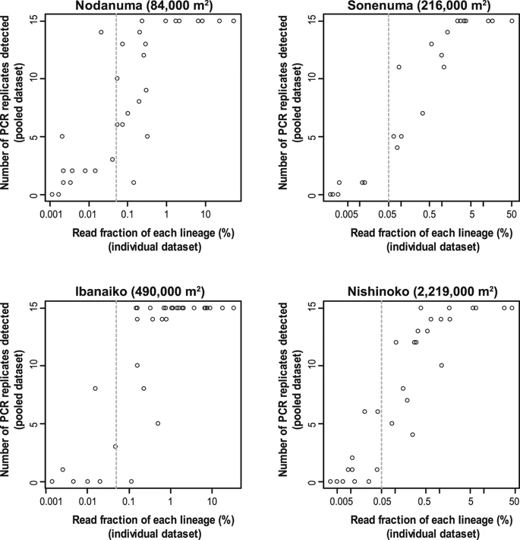
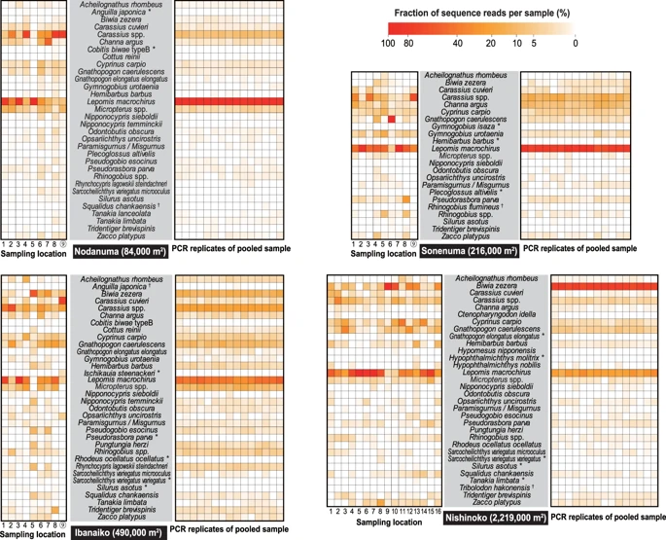
さらに、プールサンプルのPCRの繰り返し間では検出される魚類の種組成が類似していました。
下の図はNMDS(non metric multidimensional scaling)と呼ばれる地点間の群集の類似性(β多様性)を可視化する解析です(Rを使った動かし方はこちらのブログが参考になります)。
近ければ似ていて、離れていれば似ていないといった見方をします。下図上段は個別分析の魚類相のデータに対して、下段はプールサンプルについて解析を行っています。
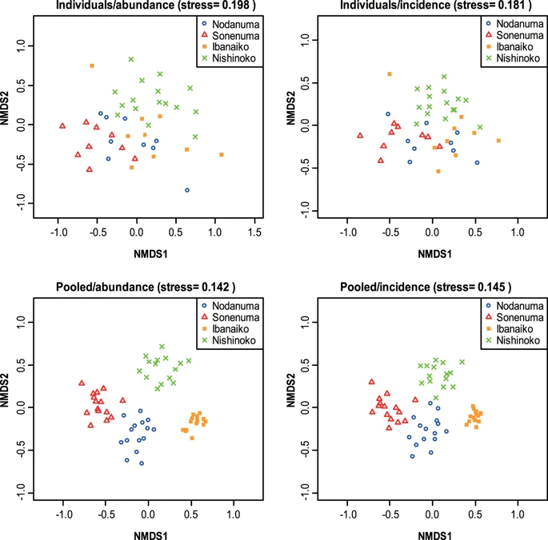
見てわかるように、個別でサンプルを分析した場合に比べて、プールしたサンプルはそれぞれの内湖ごとに点が集まっていることから、各内湖の魚類相の特徴を示していると言えます。
つまり、サンプルをプールして分析する方法は多様性検出ではなく、調査地間の種構成の特徴を比較する場合には有用である可能性があると言えます。
まとめ
- 個別のサンプル数の増加は同じ地点のサンプルをプールしてPCRの繰り返し数を増加させるよりも多くの種を検出できる
- 調査地点の特徴を比較する場合には、複数地点のサンプルをプールするほうが種構成の特徴が顕著に表れやすい
- Read数の少ない=サンプル中のDNA量が少ない種はプールしたサンプルからは検出されにくい
- あまり近くで採水すると、空間自己相関が発生する
(…可能性があるとします)
画像はジャーナルのCC BYに従う形で、記事で紹介したオープンアクセスの論文中の画像または、自身で作成したものを使用しています。