大学の時に調査した内容が論文になったので、宣伝もかねてまとめていきます。今までの系統地理研究や遺伝的多様性の調査に役立つようにと頑張りました。
目次
- 遺伝的多様性とは
- 調査する意味
- 環境DNAと遺伝的多様性研究
- 水槽水での実証研究
- 野外調査での実証研究
- 生態学研究への応用
この記事は環境DNAを使った遺伝的多様性評価法の現状について解説しています。
1~3番目は導入なので読み飛ばしてもらっても結構です。
1. 遺伝的多様性とは
遺伝的多様性は1つの生物種内の遺伝子のバリエーションの事を指します。
生物が生存するために重要な要素の1つである遺伝的多様性は、”環境の多様性” “種の多様性”とならび、3つの生物多様性の定義に含まれています。
そして、これらが維持されることで生態系のバランスが保たれています。
ただ、”遺伝子”というと遺伝情報を持つ1つのDNA領域という意味にもなるので、環境DNA分析の場合、話のベースとなるのは1塩基の多型(SNP:スニップ)のことだと思ってください。
以下の画像のように同じアユ内でも塩基の配列が異なります。

遺伝子のバリエーションともなると、私たちが普段あまり感じることのないとても小さなスケールの話しですが、生物保護の観点からは必ず押さえておく必要があります。
1種の生物でも日本各地に生息する生物はたくさんいます。その生物は同じ種だからと言って、完全に同じ(=クローン)であるとは限りません。生息地の距離が離れるにつれて遺伝的な違いが大きくなってくると思います。
それは主に気候変動や地理的隔離、周囲に生息する他の生物の違い由来すると考えられます。そういった周囲の環境変化に柔軟に対応して生存するためには遺伝レベルでの柔軟性が欠かせません。要するに遺伝的な多様性はその状況に適応し、進化するための原動力といえます。
2. 遺伝的多様性を調査する意味
遺伝的な多様性を調査すると、外見には表れない生物(集団)の状態が推定できます。
高い遺伝的な多様性を持つ集団は病気や気候変動に見舞われても、一度にすべてが死ぬことはないでしょう。ですが、遺伝的な多様性が低下すると、集団全体が同じ病気にかかったり、繁殖が成功しなかったりします。これは近交弱勢と呼ばれたりします。
また、ある閾値を超えると、元に戻れなくなるといった性質が生態学にもあります。客観的にこれは問題だと異常性を察知して、人工的に手を加えて多様性を高めようとしても、戻れない状態まで進んでしまっている場合、その種は絶滅へと向かってしまうでしょう。
このように、遺伝子レベルで多様性が失われる前に、遺伝的多様性調査によってその実態を確認するのは、生物保全において非常に重要なことだと言えます。
3. 環境DNAを用いた遺伝的多様性研究
そして、環境DNAは捕獲が許可されていない生物や捕獲が困難な場所、また広範囲を一度に調査してモニタリングを続ける場合などに特に有用な手法です。
魚、モニタリング、分布調査、多様性調査などのワードが思いついたら環境DNAを使うことを検討してみてください。
大型脊椎動物を対象とした場合、環境DNAを使った遺伝的多様性の評価論文は以下の7本です。
2016年
Population characteristics of a large whale shark aggregation inferred from seawater environmental DNA
海水中の環境DNAから推定されるジンベイザメの個体群特性
Water, water everywhere: environmental DNA can unlock population structure in elusive marine species
全ての水から:環境DNAは海洋生物の個体群構造を明らかにすることが出来る
2019年
Population-level inferences from environmental DNA-Current status and future perspectives
環境DNAから個体群レベルの推定 – 近況と将来への期待
HaCeD-Seq: a Novel Method for Reliable and Easy Estimation About the Fish Population Using Haplotype Count from eDNA
HaCaD-Seq:環境DNAのハプロタイプ数を用いた魚類個体群の簡単で信頼できる革新的な推定方法
Evaluating intraspecific genetic diversity using environmental DNA and denoising approach: A case study using tank water
水槽水での実証研究: 環境DNAを用いた遺伝的多様性の評価とデノイジングアプローチ
2020年
Environmental DNA analysis shows high potential as a tool for estimating intraspecific genetic diversity in a wild fish population
環境DNA分析は魚類の野外個体群の遺伝的多様性を推定するためのツールとして高い可能性を示す
Quantitative evaluation of intraspecific genetic diversity in a natural fish population using environmental DNA analysis
自然個体群における環境DNAを用いた遺伝的多様性の定量的な評価法
このようにいろいろハプロタイプ推定に関する論文は出ていますが、環境DNAでハプロタイプを検出する時に最も重要な事はデノイズです。
1塩基の違い一つ一つをハプロタイプとするので、ノイズ=“PCRエラーやNGSの読み取りミス”を取り除く処理であるデノイズの有無で結果が大きく異なります。
分かりやすいのがこの論文です。
4. 水槽水での実証研究
水槽水での実証研究 : 環境DNAを用いた遺伝的多様性の評価とデノイジングアプローチ
Evaluating intraspecific genetic diversity using environmental DNA and denoising approach: A case study using tank water
ざっくりとしたまとめは以下の通りです。
水槽実験において、飼育個体に由来する複数のアユmtDNAハプロタイプを環境DNA分析により同時に検出できたという内容。環境DNA分析を用いた遺伝的多様性検出における最大の問題点であるHTSデータ中のPCRやシーケンスエラーに由来する偽陽性ハプロタイプの99%を、「ASV法によるデノイズ処理+複数のPCR反復間における出現頻度に基づくデータ選択」という新しい解析アプローチにより排除し、検出精度を大幅に改善することに成功しました。
著者HP -辻冴月Web Site-
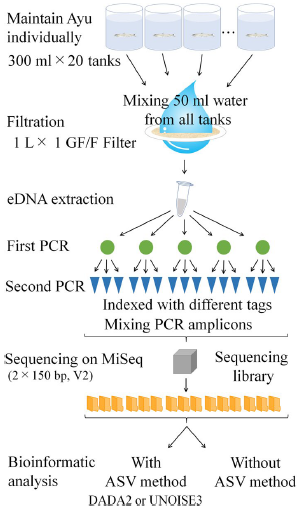
1st PCRを各サンプル5繰り返しで分析し、それぞれの繰り返しごとに2nd PCRを3繰り返し、1つの水槽から合計15繰り返しを生成しています。
飼育実験に使用した20個体のうち研究に用いたプライマー領域のハプロタイプ数は9ハプロタイプでした。
結果として、9の飼育個体由来のハプロタイプの他に検出されるはずのない5683のハプロタイプ(=偽陽性ハプロタイプ)が検出されました。
ここにDADA2とUNOISE3と呼ばれる、統計的にエラー配列を修正・除去するアルゴリズムを適用すると以下のようになります。
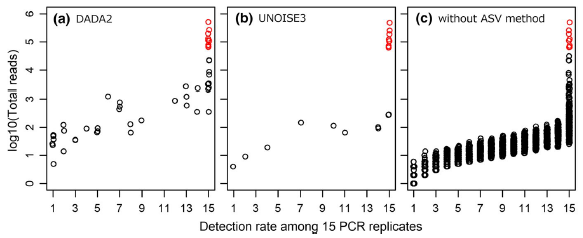
右図( 赤:飼育個体ハプロタイプ 黒:偽陽性と考えられるハプロタイプ )
デノイジングアプローチを加えることにより、偽陽性と考えられるハプロタイプ(黒丸)が分かりやすく激減しています。また、正しいとされるハプロタイプは必ず15繰り返し全てで、どの偽陽性ハプロタイプのリードよりも多く検出されたそうです。
5. 野外調査での実証研究
環境DNA分析は魚類の野外個体群の遺伝的多様性を推定するためのツールとして高い可能性を示す
Environmental DNA analysis shows high potential as a tool for estimating intraspecific genetic diversity in a wild fish population
水槽実験で試料中に含まれる既知のハプロタイプを環境DNAで検出できるか検討したので、次は河川での捕獲調査と環境DNAでの結果を比較します。
オープンアクセスではないので、内容の公開は控えめにアブストベースで紹介していきます。
ざっくりとしたまとめは以下の通りです。
環境DNA分析を用いて野外アユ集団内の遺伝的多様性をどれだけちゃんと検出できるか?を捕獲調査の結果と比較することで検証した内容。結果として、環境DNA分析は大規模な捕獲調査(約100尾/地点)と同等、もしくはそれ以上の検出力を持つことが確認されました。捕獲調査のように、長い配列を使った解析はできませんが、多地点・高頻度で遺伝的多様性のモニタリングやスクリーニングをする際には、費用対効果・網羅性の面で環境DNA分析がうってつけの方法であると思います。
著者HP -辻冴月Web Site-
サンプリングは同日に河川から水試料(環境DNA用)と組織サンプル(サンガーシーケンスで分析用)を採取しています。
環境DNAは10Lの水試料を20枚のフィルターで均等な量でろ過して、それぞれを15回のPCR繰り返しで分析しました。組織サンプルは96個体のアユをサンガーシーケンスにて各個体の配列を決定しました。環境DNAの解析時のデノイズは水槽実験でも使っていたDADA2とUNOISE3を使用しています。
結果として、96個体のサンガーシーケンスから42ハプロタイプが検出されました。それらのハプロタイプのうち、環境DNAの水試料からUNOSE3では38ハプロタイプが、DADA2では41ハプロタイプが検出されました。また、DADA2をデノイズ法として用いた場合、1つのハプロタイプを除いて、少なくとも2/96個体以上が有するハプロタイプがすべてのフィルター反復から検出され、15回の繰り返しの検出確率が98%以上でした。
これらのことから、環境DNAを用いた遺伝的多様性調査の場合、500mLの水をろ過したGF/Fフィルター1枚から2/96個体しか保有しないようなかなり稀なハプロタイプまで検出できる可能性があるということが言えると思います。また、論文の実験のように1サンプルにつきフィルターを20枚を使用することは現実的ではなく、論文内では通常の遺伝的多様性調査で分析されるであろう個体数以上のハプロタイプが検出することが望めるPCRの反復数とフィルター数を考察したりもしています。(お水を500mL×複数すくって分析すればかなりの網羅性を望めます。。ごにょごにょ)
6. 生態学研究への応用
自然個体群における環境DNAを用いた遺伝的多様性の定量的な評価法
Quantitative evaluation of intraspecific genetic diversity in a natural fish population using environmental DNA analysis. Molecular Ecology Resources.
上記の論文で、捕獲調査と比較してかなり稀なハプロタイプまで環境DNAで検出できることが分かりました。
また、Ushio et al.2018で定量的な環境DNAメタバーコーディング法が提案されているので、その原理を応用して”自然個体群における環境DNAを用いた遺伝的多様性の定量的な評価法”の検証をしました。
ざっくりとしたまとめは以下の通りです。
環境DNA分析を用いて、遺伝的多様性を量的に評価することが可能になりました。また、「どんなハプロタイプがあるか?」だけでなく、「各ハプロタイプがどれだけあるか?」を評価できるようになったことで、各ハプロタイプのコピー数を個体数の代用とし、塩基多様度や有効集団サイズの計算ができるようになったことを報告しています。これにより、新たに環境DNA分析の集団遺伝学や系統地理への利用の道が拓かれたと言えます。
著者HP -辻冴月Web Site-
端的に内容が示されているので、あまり追記することがありません。笑
まとめ
デノイズ法や実際の飼育環境と野外調査での検証により、環境DNA使ったハプロタイプ推定はかなり実用的なレベルまできていると思います。うまく使えば、今まで行われてきたような集団遺伝学や系統地理の一つの調査法になりえるかもしれません。
1杯の水から、どんなハプロタイプが出現するかを同地点で追っていくのも面白いと思いますし、定量的な評価を加えることで、集団構成の変化や場所による比較を様々な種で行うこともできるようになっていくのではないでしょうか。
※毎回解説の書き方が変わってますが、分かりやすい形を模索中です。ご了承ください。






