魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish : マイフィッシュ)の開発者本人によるレビュー論文の和訳第二弾です。まだ、数パート続きます。
研究課題を考える学生さんや新しく始める人などの仕事や研究の手助けになれば嬉しいです@しばた
MiFishメタバーコーディング:環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ
MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
この記事の内容
目次
- Introduction
- 競合するUniversal PCRプライマーとの性能比較
- MiFishプライマーの分類学的最適化
- 望ましい実験室設定と改訂された実験プロトコルについて
- 新しいバイオインフォマティクスパイプラインと参照データベース(今回はここ)
- さまざまな水生環境での実証研究のレビュー
- 海の魚類群集
- 淡水の魚類群集
- 河口域の魚類群集
- MiFishプライマーを用いた新しい技術の開発
- 他の生物へのMiFishメタバーコーディングの応用
- バルクDNAサンプルへのMiFishメタバーコーディングの応用
- さいごに
- 感想
1,2章の内容はこちら
3章の内容はこちら
4章の内容はこちら
5. 新しいバイオインフォマティクスパイプラインと参照データベース
MiFishメタバーコーディングでは、ハイスループットシーケンサーを用いた塩基配列決定により、一晩で数千万から数十億のDNA配列(リード)が出力されます。
よって、大量の生の配列(リード)を前処理して解析するためには、バイオインフォマティクスパイプラインが不可欠です。
MiFishパイプラインの利点と欠点
Miya et al. 2015は、利用可能な様々ないくつかのソフトウェアを組み合わせたバイオインフォマティクスパイプラインを構築しました。
このパイプラインはその後、MitoFishサーバー上に実装され、現在はウェブサイトで利用できるようになっています(Sato et al. 2018)
しかしながら、ハイスループットシーケンサーから出力されるデータには、ランダムな位置で1塩基または数塩基、真の生物学的配列と異なる多数の偽の配列が含まれていることがよく知られています(Coissac et al.2012, Edgar 2016)。
これらのミスは、1st, 2nd PCRやMiSeqシークエンシング時のクラスター増幅、また、配列解読の際に酵素が誤った塩基を組み込んだときに生じる可能性があります。
さらに、MiSeqシーケンシングシステム中の画像解析も、約0.1〜1%の塩基にエラーが含まれている場合があります(Fox et al. 2014)。
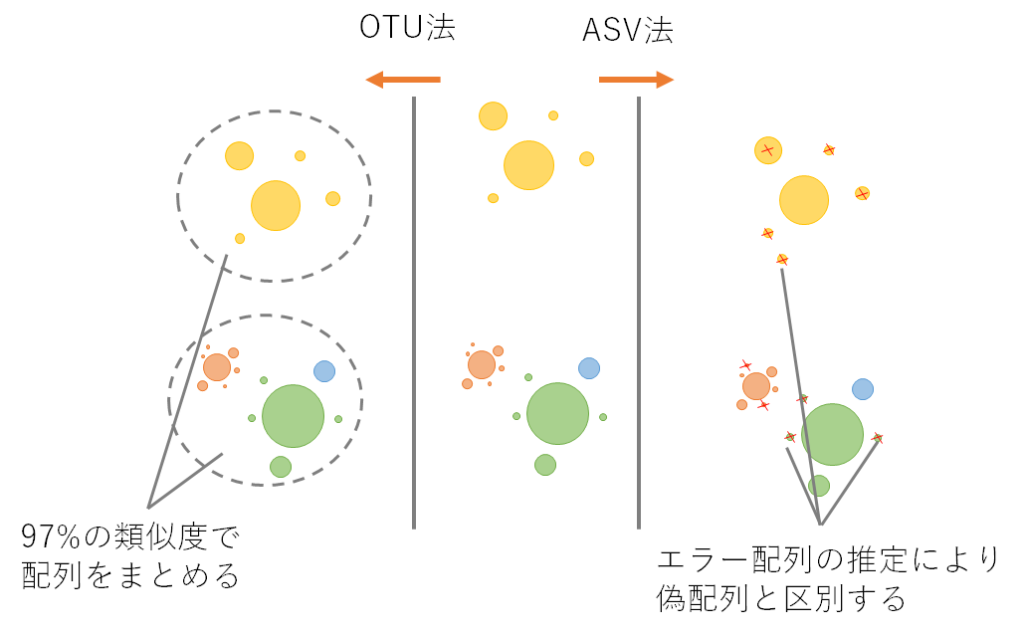
ウェブ上で利用可能なMiFishパイプラインは、そのような誤った配列を明示的に除去しない代わりに、固定の閾値 (例えば、97%の類似度) で類似している配列のクラスタリングを行います(Callahan et al. 2016)。
このように、得られた配列を分類するために任意の類似度でまとめられた配列をOTUといい、OTUへのDNA配列のクラスタリングはOTU法と呼ばれます。
その出力は再利用可能性、再現性、および包括性を欠くことが実証されています (Callahan et al.2017)。
OUT法とは別の配列推定法
近年、微生物学の分野では、生物学的配列のより正確な推定のためにOTU法に代わって、ASV法が開発されました。
ASV法は増幅や配列決定エラーが導入される前のサンプル中の生物学的な配列を推定し、1塩基違いの配列を区別します(Callahan et al. 2017)。
ASV法の中心となるプロセスは、前者の方が後者よりも繰り返し観測される可能性が高いという期待に基づいて、生物学的配列と誤配列を区別する「デノイジング」と呼ばれるものです(Tsuji et al. 2020)。
ASV法を組み込んだ新しいパイプライン(PMiFish)
我々の研究グループでは現在、このノイズ除去処理をカスタムバイオインフォマティクスパイプラインに組み込んでおり、生データの前処理からカスタムデータベースに基づく分類学的割り当てまでのパイプラインを更新したものをまとめています(PMiFish ver. 2.4; 最新版はhttps://github.com/rogotoh/PMiFishから入手可能)。
なお、PMiFishパイプラインのほとんどのパラメーター(例えば、分類学的割り当てにおける配列同一性の割合など)は、目的に応じてユーザーが変更できるようになっています。
MiSeqから出力されたリードのデータ前処理と解析は、USEARCH v10.0.24 (Edgar 2010) を使用して、以下のステップに従って実行されます。
- 品質フィルタリングされたフォワード(R1)とリバース(R2)リードの組み立て
- プライマー配列の除去
- クオリティーフィルタリング
- 重複の除去
- ノイズ配列の除去
- 生物種名の割り当て
Edgar 2010は、誤配列の主な原因となるシングルトン、ダブルトン、トリプレトン (全配列の中で1 or 2 or 3リードしかない配列) の除去を推奨していることに注意しましょう。
分類学的割り当ての後、PMiFish ver. 2.4では、検出されたMiFish配列と、それらのファミリーからの参照配列(カスタムデータベースに格納されている)からなるファミリーレベルの系統図を生成することができます。
このようなファミリーレベルの系統を視覚的に確認することで、分類学的割り当ての信憑性を確認することができます。
バイオインフォマティクスパイプラインと系統に基づく確認についての詳細は、Komai et al.2018かOka et al. 2020を参照してください。
参照データベースについて
Miya et al. 2015は魚類の多様性調査に関する主流の方法となる前に環境DNAメタバーコーディングに基づくアプローチはいくつかの解決すべき方法論的な課題があると指摘しています。
そのうち一つの課題は、リファレンスの完全性と正確性です。
世界中の水生環境から知られている32,000種以上の魚類を含む膨大な多様性を考慮すると、現在のデータベースは満足のいくものではありません。
実際、カスタム参照配列データベース (“MiFish DB”と命名) には、原著論文が発表された2014年10月4日時点で、457科1827属に分類された約4230種の魚種をカバーする5085本の配列が含まれていました。
その後、MiFish DBは過去5年間で着実に検出できる分類群を拡大し、2020年6月14日時点で、登録配列数は約2倍の9708本となりました(これは62目、479科、2766属に属する約8375種の魚類をカバーしていることになります(Fig13))。
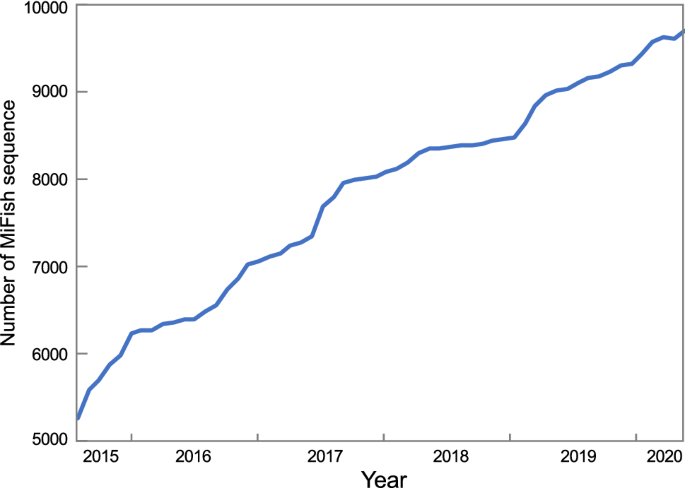
FishBase (Froese and Pauly 2019) の種の多様性に関する情報によると、全世界の魚種数は約34,152種、62目、515科、5124属に分類されています。
現在のMiFish DB ver.38は全世界の魚類を目:100%、科:90.3%、属:54.0%、種:24.5%網羅しています。
日本の魚類は、これまでに4554種が記録されており、MiFish DB ver. 38では、50目(100%)、345科(90.6%)、1302属(81.2%)、3257種(71.5%)をカバーしています。
これらのカバー率は、検出された配列の正確な分類学的割り当てには程遠いものであり、国際共同研究によるデータベース構築が望まれます。
この点に関しては、カリフォルニア海流地域の関連大学・研究機関の共同イニシアティブにより、MiFishメタバーコーディングを強化するための独自の参照データベースが構築され、同地域の既知種864種のうち717種の参照配列が構築されていることは注目すべき点です。
第4章 和訳おわり
Reference
- Edgar 2010
Search and clustering orders of magnitude faster than BLAST - Coissac et al.2012
Bioinformatic challenges for DNA metabarcoding of plants and animals - Fox et al. 2014
Accuracy of next generation sequencing platforms - Miya et al.2015
MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species - Edgar 2016
UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing - Callahan et al.2016
DADA2: high-resolution sample inference from Illumina amplicon data - Callahan et al. 2017
Exact sequence variants should replace operational taxonomic units in marker-gene data analysis - Sato et al. 2018
MitoFish and MiFish pipeline: a mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding - Froese and Pauly 2019
Fish base - Komai et al. 2018
Development of a new set of PCR primers for eDNA metabarcoding decapod crustaceans - Oka et al.2020
Environmental DNA metabarcoding for biodiversity monitoring of a highly-diverse tropical fish community in a coral-reef lagoon: Estimation of species richness and detection of habitat segregation - Tsuji et al.2020
Evaluating intraspecific genetic diversity using environmental DNA and denoising approach: a case study using tank water